Genska regulatorna mreža



Genska (ili genetička) regulatorna mreža (GRN) je skup molekulskih regulatora koji međusobno djeluju i s drugim supstancama u ćeliji da upravlja eekspresijom gena, nivoima iRNK i proteina koji, zauzvrat, određuju funkciju ćelije. GRN takođe ima centralnu ulogu u morfogenezi, stvaranju tjelesnih struktura, što je centralno za evolucijsku razvojnu biologiju (evo-devo).
Regulator može biti DNK, RNK, protein ili bilo koja kombinacija dva ili više od ova tri koja formiraju kompleks, kao što je specifična sekvenca DNK i transkripcijski faktor za aktiviranje te sekvence. Interakcija može biti direktna ili indirektna (preko transkribovane RNK ili prevedenog proteina). Općenito, svaka molekula iRNK nastavlja da proizvodi određeni protein (ili skup proteina). U nekim slučajevima ovaj protein će biti strukturni, i akumuliraće se na ćelijskoj membrani ili unutar ćelije da bi joj dao određena strukturna svojstva. U drugim slučajevima protein će biti enzim, tj. mikromašina koja katalizuje određenu reakciju, kao što je razgradnja izvora hrane ili toksina. Neki proteini ipak služe samo za aktiviranje drugih gena, a to su transkripcijski faktoru koji su glavni u regulatornim mrežama ili kaskadama. Vezivanjem za promotorski region na početku drugih gena oni ih uključuju, pokrećući proizvodnju drugog proteina, i tako dalje. Neki faktori transkripcije su inhibitorni.[1]
Kod jednoćelijskih organizama, regulatorne mreže reaguju na vanjsko okruženje, optimizirajući ćeliju u datom trenutku za preživljavanje u ovom okruženju. Tako će ćelija kvasca, koja se nađe u rastvoru šećera, uključiti gene da stvaraju enzime koji prerađuju šećer u alkohol. Ovaj proces, koji se povezuje sa proizvodnjom vina, je način na koji ćelija kvasca živi, dobijajući energiju za razmnožavanje, što bi u normalnim okolnostima povećalo izglede za njen opstanak.
Kod višećelijskih životinja isti princip je stavljen u službu genskih kaskada koje kontroliraju oblik tijela.[2] Svaki put kada se ćelija podijeli, nastaju dvije ćelije koje, iako sadrže isti genom u potpunosti, mogu se razlikovati po tome koji su geni uključeni i stvaraju proteine. Ponekad 'samoodrživa povratna sprega' osigurava da ćelija zadrži svoj identitet i da ga prenosi dalje. Manje je shvaćen epigenetički mehanizam kojim modifikacija hromatina može obezbijediti ćelijsku memoriju, blokiranjem ili omogućavanjem transkripcije. Glavna karakteristika višećelijskih životinja je upotreba gradijenata morfogena, koji u stvari obezbjeđuju sistem pozicioniranja koji ćeliji određuje gdje se nalazi u tijelu, a time i kakva će ćelija postati. Gen koji je uključen u jednoj ćeliji može napraviti proizvod koji napušta ćeliju i difundira kroz susjedne ćelije, ulazi u njih i uključuje gene samo kada je prisutan iznad određenog nivoa praga. Ove ćelije se na taj način induciraju u novu sudbinu i mogu čak generirati druge morfogene koji signaliziraju natrag izvornoj ćeliji. Na većim udaljenostima, morfogeni mogu koristiti aktivni proces transdukcija signala. Takva signalizacija kontrolira embriogenezu, izgradnju plana građe od nule kroz niz uzastopnih koraka. Oni također kontroliraju i održavaju tijela odraslih putem povratnih informacija procesa, a gubitak takve povratne informacije zbog mutacije može biti odgovoran za proliferaciju čelija koja se vidi kod kancera. Paralelno sa ovim procesom izgradnje strukture, kaskada gena uključuje gene koji čine strukturne proteine koji svakoj ćeliji daju fizička svojstva koja su joj potrebna.
Pregled
[uredi | uredi izvor]Na jednom nivou, biološke ćelije se mogu smatrati "delimično pomješanim vrećama" bioloških hemikalija - u raspravi o mrežama regulacije gena, ove hemikalije su uglavnom informacijska RNK (iRNK) i proteini koje nastaju ekspresijom gena. Ove iRNK i proteini međusobno djeluju s različitim stupnjevima specifičnosti. Neki se šire oko ćelije. Drugi su vezani za ćelijsku membranu, u interakciji sa molekulama u okolini. Drugi prolaze kroz ćelijske membrane i posreduju signale velikog dometa drugim ćelijama u višećelijskom organizmu. Ove molekule i njihove interakcije čine „mrežu regulacije gena“. Tipska regulatorna mreža gena izgleda otprilike ovako:
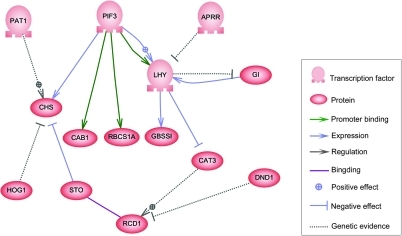
Čvorovi ove mreže mogu predstavljati gene, proteine, iRNK, proteinske/proteinske komplekse ili ćelijske procese. Čvorovi koji su prikazani kako leže duž vertikalnih linija povezani su sa interfejsima ćelija/okruženje, dok su ostali slobodno plutajući i mogu difundirati. Rubovi između čvorova predstavljaju interakcije između čvorova, koje mogu odgovarati pojedinačnim molekulskim reakcijama između DNK, iRNK, miRNK, proteina ili molekulskih procesa kroz koje proizvodi jednog gena utiču na proizvode drugog, iako nedostatak eksperimentalno dobijenih informacija često implicira da neki reakcije nisu modelirane na tako finom nivou detalja. Ove interakcije mogu biti induktivne (obično predstavljene vrhovima strelica ili znakom +), s povećanjem koncentracije jedne koja dovodi do povećanja druge, inhibitorne (predstavljene popunjenim krugovima, tupim strelicama ili znakom minus), s povećanjem u jednom dovodi do smanjenja u drugom, ili dvostruko, kada ovisno o okolnostima regulator može aktivirati ili inhibirati ciljni čvor. Čvorovi se mogu regulisati direktno ili indirektno, stvarajući povratne petlje, koje formiraju ciklične lance u zavisnosti u topološkoj mreži. Mrežna struktura je apstrakcija molekulske ili hemijske dinamike sistema, koja opisuje mnogostruke načine na koje jedna supstanca utiče na sve druge sa kojima je povezana. U praksi, takvi GRN su zaključeni iz biološke literature o datom sistemu i predstavljaju destilat kolektivnog znanja o skupu srodnih biohemijskih reakcija. Da bi se ubrzalo ručno određivanje GRN-ova, neki nedavni napori pokušavaju koristiti tekstualno rudarenje, kurirane baze podataka, mrežno zaključivanje iz masivnih podataka, provjeru modela i druge tehnologije za ekstrakciju informacija u tu svrhu.[3]
Geni se mogu posmatrati kao čvorovi u mreži, pri čemu su ulaz proteini kao što su transkripcijski faktori, a izlaz je nivo ekspresije gena. Vrijednost čvora ovisi o funkciji koja ovisi o vrijednosti njenih regulatora u prethodnim vremenskim koracima (u dolje opisanoj Booleovoj mreži to su Booleove funkcije, tipski AND, OR i NOT). Ove funkcije se tumače kao obavljanje neke vrste obrada informacija unutar ćelije, koja određuje ćelijsko ponašanje. Osnovni pokretači unutar ćelija su koncentracije nekih proteina, koji određuju kako prostorne (lokacija unutar ćelije ili tkiva) tako i vremenske (ćelijski ciklus ili razvojna faza) koordinate ćelije, kao svojevrsne "ćelijske memorije". Mreže gena tek počinju da se shvataju, a sljedeći korak za biologiju je da pokuša da izvede funkcije za svaki "čvor" gena, kako bi se razumelo ponašanje sistema u rastućim nivoima složenosti, od gena do signalnog puta, nivoa ćelije ili tkiva.[4]
Matematički modeli GRN-a su razvijeni da bi se uhvatilo ponašanje sistema koji se modelira, a u nekim slučajevima i generirala predviđanja koja odgovaraju eksperimentalnim zapažanjima. U nekim drugim slučajevima pokazalo se da modeli daju tačna nova predviđanja, koja se mogu eksperimentalno testirati, sugerirajući tako nove pristupe za istraživanje u eksperimentu koji se ponekad ne bi uzeti u obzir u dizajnu protokola eksperimentalne laboratorije. Tehnike modeliranja uključuju diferencijakne jednačine (ODE), Boleove mreže, petočlana mreža, Bayesova mreža, grafički model Gaussove mreže, stohastički i procesni računi.[5] Suprotno tome, predložene su tehnike za generiranje modela GRN-ova koji najbolje objašnjavaju skup vremenskih serija opservacija. Nedavno je pokazano da signal modifikacije histona ChIP-seq ima više korelacija sa motivima transkripcijskog faktora na promotorima u poređenju sa nivoom RNK.[6] Stoga se predlaže da bi modifikacija histona vremenske serije ChIP-seq mogla pružiti pouzdaniji zaključak o mrežama regulacije gena u poređenju sa metodama zasnovanim na nivoima ekspresije.
Modeliranje
[uredi | uredi izvor]Spojene obične diferencijalne jednadžbe
[uredi | uredi izvor]Uobičajeno je modelirati takvu mrežu sa skupom spregnutih običnih diferencijalnih jednačina (ODE) ili SDE, koji opisuju kinetiku reakcije sastavnih dijelova. Pretpostavimo da data regulatorna mreža ima čvorova i neka predstavlja koncentracije odgovarajućih supstanci u trenutku . Tada se vremenska evolucija sistema može približno opisati sa:
Gdje funkcija izraziti ovisnost o koncentracijama drugih supstanci prisutnih u ćeliji. Funkcije su u konačnici izvedene iz osnovnih principa hemijske kinetike ili jednostavnih izraza izvedenih iz ovih npr. Michaelis-Mentenova enzimska kinetika. Stoga se funkcionalni oblici obično biraju kao polinomi nižeg reda ili Hilova funkcija koji služe kao osnovsa za pravu molekusku dinamiku. Takvi modeli se zatim proučavaju pomoću matematike nelinearne dinamike. Informacije specifične za sistem, kao što su konstante brzina reakcije i osjetljivosti, kodirane su kao konstantni parametri.[7]
Rješavanjem za fiksnu tačku sistema:
za sve , dobijamo (moguće nekoliko) koncentracijske profile proteina i iRNK koji su teorijski održivi (iako ne nužno stabilni). Stabilno stanje kinetičkih jednačina tako odgovaraju potencijalnim tipovima ćelija, a oscilacijsko rješenja gornje jednačine za prirodno ciklične tipove ćelija. Matematička stabilnost ovih atraktora se obično može okarakterisati predznakom viših derivata u kritičnim tačkama, a zatim odgovarati biohemijska stabilnost profila koncentracije. Kritične tačke i bifurkacija u jednačinama odgovaraju kritičnim stanjima ćelije u kojima male perturbacije stanja ili parametara mogu prebaciti sistem između jedne od nekoliko stabilnih diferencijacijskih sudbina. Trajektorije odgovaraju odvijanju bioloških puteva i tranzijenta jednadžbi prema kratkoročnim biološkim događajima.
Također pogledajte
[uredi | uredi izvor]- Plan tijela
- Cis-regulatorni modul
- Genenetwork (baza podataka)
- Morfogen
- Operon
- Sinekspresija
- Sistemska biologija
- Ponderisana analiza mreže koekspresije gena
Reference
[uredi | uredi izvor]- ^ Latchman DS (septembar 1996). "Inhibitory transcription factors". The International Journal of Biochemistry & Cell Biology. 28 (9): 965–974. doi:10.1016/1357-2725(96)00039-8. PMID 8930119.
- ^ Davidson E, Levin M (april 2005). "Gene regulatory networks". Proceedings of the National Academy of Sciences of the United States of America. 102 (14): 4935. Bibcode:2005PNAS..102.4935D. doi:10.1073/pnas.0502024102. PMC 556010. PMID 15809445.
- ^ Leitner F, Krallinger M, Tripathi S, Kuiper M, Lægreid A, Valencia A (juli 2013). "Mining cis-regulatory transcription networks from literature". Proceedings of BioLINK SIG 2013: 5–12.
- ^ Azpeitia E, Muñoz S, González-Tokman D, Martínez-Sánchez ME, Weinstein N, Naldi A, et al. (februar 2017). "The combination of the functionalities of feedback circuits is determinant for the attractors' number and size in pathway-like Boolean networks". Scientific Reports. 7: 42023. Bibcode:2017NatSR...742023A. doi:10.1038/srep42023. PMC 5301197. PMID 28186191.
- ^ Banf M, Rhee SY (januar 2017). "Computational inference of gene regulatory networks: Approaches, limitations and opportunities". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1860 (1): 41–52. doi:10.1016/j.bbagrm.2016.09.003. PMID 27641093.
- ^ Kumar V, Muratani M, Rayan NA, Kraus P, Lufkin T, Ng HH, Prabhakar S (juli 2013). "Uniform, optimal signal processing of mapped deep-sequencing data". Nature Biotechnology. 31 (7): 615–622. doi:10.1038/nbt.2596. PMID 23770639.
- ^ Chu D, Zabet NR, Mitavskiy B (april 2009). "Models of transcription factor binding: sensitivity of activation functions to model assumptions" (PDF). Journal of Theoretical Biology. 257 (3): 419–429. Bibcode:2009JThBi.257..419C. doi:10.1016/j.jtbi.2008.11.026. PMID 19121637.
Dopunska literatura
[uredi | uredi izvor]- Bolouri H, Bower JM (2001). Computational modeling of genetic and biochemical networks. Cambridge, Mass: MIT Press. ISBN 978-0-262-02481-5.
- Kauffman SA (mart 1969). "Metabolic stability and epigenesis in randomly constructed genetic nets". Journal of Theoretical Biology. 22 (3): 437–467. Bibcode:1969JThBi..22..437K. doi:10.1016/0022-5193(69)90015-0. PMID 5803332.
Vanjski linkovi
[uredi | uredi izvor]- Plant Transcription Factor Database and Plant Transcriptional Regulation Data and Analysis Platform Arhivirano 13. 5. 2020. na Wayback Machine
- Open source web service for GRN analysis
- BIB: Yeast Biological Interaction Browser Arhivirano 30. 9. 2011. na Wayback Machine
- Graphical Gaussian models for genome data – Inference of gene association networks with GGMs
- A bibliography on learning causal networks of gene interactions – regularly updated, contains hundreds of links to papers from bioinformatics, statistics, machine learning.
- https://web.archive.org/web/20060907074456/http://mips.gsf.de/proj/biorel/ BIOREL is a web-based resource for quantitative estimation of the gene network bias in relation to available database information about gene activity/function/properties/associations/interactio.
- Evolving Biological Clocks using Genetic Regulatory Networks – Information page with model source code and Java applet.
- Engineered Gene Networks
- Tutorial: Genetic Algorithms and their Application to the Artificial Evolution of Genetic Regulatory Networks
- BEN: a web-based resource for exploring the connections between genes, diseases, and other biomedical entities
- Global protein-protein interaction and gene regulation network of Arabidopsis thaliana Arhivirano 16. 3. 2016. na Wayback Machine







