Chromosomenterritorium
 Van Wikipedia, de gratis encyclopedie
Van Wikipedia, de gratis encyclopedie
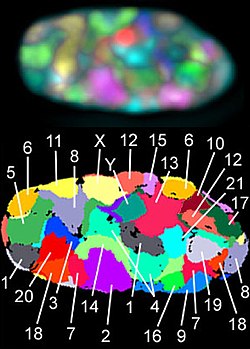
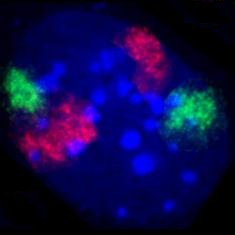
Ein Chromosomenterritorium ist der Bereich innerhalb eines Zellkerns, der von einem Chromosom in der Interphase eingenommen wird. Chromosomenterritorien haben variable Formen, die sich nicht nur zwischen verschiedenen Chromosomen, sondern auch für dasselbe Chromosom von Kern zu Kern unterscheiden. Es handelt sich nicht um feste Gebilde; andere Moleküle (wie Proteine) können durch sie hindurch diffundieren. Auch die Arme von Chromosomen sowie einzelne Chromosomenbanden bilden abgegrenzte Bereiche.[1] Chromosomenterritorien sind dynamische Strukturen, deren Gene bei Einsetzen der Transkription verschoben werden können.[2]
Geschichte[Bearbeiten | Quelltext bearbeiten]
Bereits in den 1880er Jahren hatte Carl Rabl erstmals vorgeschlagen, dass sich Chromosomen zwischen zwei Kernteilungen (Mitose) nicht vermischen, sondern auch im Zellkern der Interphase voneinander getrennt sind. Er schlug für den Bereich eines Chromosoms den Begriff Territorium vor. Rabls diesbezügliche Ansichten wurden von Theodor Boveri am Anfang des 20. Jahrhunderts unterstützt. Als im späteren Verlauf des 20. Jahrhunderts die Elektronenmikroskopie Einblicke in biologische Präparate von zuvor ungekannter Auflösung erlaubte, war es nicht möglich, im Zellkern Grenzen zwischen dem Chromatin einzelner Chromosomen zu beobachten. Daher setzte sich zunehmend die Ansicht durch, dass sich Chromatinfäden der verschiedenen Chromosomen innerhalb des Zellkerns stark vermischen, ähnlich wie lange Spaghetti auf einem Teller.
Hinweise, dass Chromosomen doch Territorien bilden, ergaben sich in den 1970er Jahren aus Experimenten, in denen Zellkerne mit UV-Lasern bestrahlt wurden.[3] Bei diesen Versuchen wurde jeweils nur ein kleiner Teil des Zellkerns lebender Zellen bestrahlt. Wären Chromosomen tatsächlich wie Spaghetti im ganzen Kern verteilt, so hätten bei der Bestrahlung alle oder doch zumindest die meisten Chromosomen getroffen werden müssen. In der auf die Bestrahlung folgenden Mitose war jedoch nachweisbar, dass nur wenige Chromosomen Strahlenschäden aufwiesen, die Mehrheit aber unversehrt war. Dies sprach für eine regional begrenzte Ausbreitung der Chromosomen im Zellkern. Ein direktes Sichtbarmachen von Chromosomenterritorien gelang etwa zehn Jahre später mit Hilfe der Fluoreszenz-in-situ-Hybridisierung[4][5] (Siehe auch Abbildungen).
Vorkommen[Bearbeiten | Quelltext bearbeiten]
Der Nachweis von Chromosomenterritorien erfordert, dass Sonden für eine Fluoreszenz-in-situ-Hybridisierung verfügbar sind, die genau die DNA eines Chromosoms enthält. Derartige Sonden sind aufwändig herzustellen und daher nur für einige Arten verfügbar. Chromosomenterritorien wurden in Zellkernen des Menschen und aller untersuchten Tiere und Pflanzen gefunden. Zu den untersuchten Arten gehören etliche Primaten, Mäuse (Hausmaus und andere) und das Haushuhn. Bei Pflanzen war der Nachweis schwierig, da lange Zeit keine geeigneten Sonden zur Verfügung standen. Auch hier ließen sich Chromosomenterritorien letztlich nachweisen, beispielsweise in der häufig für Versuche verwendeten Ackerschmalwand (Arabidopsis thaliana)[6].
Die weitere Verbreitung bei den Eukaryoten ist nicht sicher. In Organismen mit kleinen Chromosomen, wie etwa der Hefe Saccharomyces cerevisiae, ist ein Nachweis aufgrund der geringen Chromosomengröße und der begrenzten Auflösung des Lichtmikroskops kaum möglich. Es ist denkbar, dass bei solch kleinen Chromosomen eine Durchmischung der Chromatinfäden verschiedener Chromosomen stattfindet und es nicht zur Ausbildung von Territorien kommt.
Anordnung[Bearbeiten | Quelltext bearbeiten]
Die Anordnung der Chromosomenterritorien im Zellkern ist sehr variabel. Wenn jedoch viele Zellkerne untersucht werden, lässt sich feststellen, dass bestimmte Muster häufiger auftreten als andere. Die Territorien genreicher Chromosomen sind häufiger in der Mitte des Kerns zu finden, als solche genarmer Territorien.[7] Solch eine nicht zufällige radiale Verteilung der Territorien wurde beispielsweise in menschlichen Lymphozyten beschrieben, in denen die ähnlich großen, aber sehr genreichen beziehungsweise sehr genarmen Chromosomen 18 und 19 untersucht wurden.[1] Diese Beobachtungen einer radialen Anordnung im Kern abhängig von der Gendichte wurde durch Analysen bestätigt, die alle menschlichen Chromosomen umfassen.[8] Es gibt jedoch ausgeprägte Zell-zu-Zell-Variationen von Nachbarschaftsanordnungen dieser Territorien in allen bisher untersuchten Zelltypen.[8] Diese Gendichte-abhängige Verteilung von Chromosomenterritorien wurde auch in Zellkernen von Mäusen gefunden,[1] so dass sie über mindestens eine Zeitspanne von 60 Millionen Jahre konserviert wurde. Dies spricht für einen signifikanten Vorteil dieser Anordnung.[7]
Die Frage nach der biologischen Bedeutung dieser Anordnung versucht die „Bodyguard“-Hypothese von T.C. Hsu zu beantworten. Sie besagt, dass genreiches Material im Zellkerninneren vor Schädigungen von außen geschützt ist. Hsu ging davon aus, dass genarmes, konstitutives peripheres Heterochromatin das Euchromatin vor chemischen Mutagenen und Röntgenstrahlung schützen kann, da seine Position während der Interphase angrenzend an die Kernmembran liegt. Es ist daher so positioniert, dass es mehr Umweltrisiken absorbiert als zentrales Euchromatin und kann so die DNA dort vor Schäden und Mutationen schützen.[7][9] Die genarmen Chromosomen-Abschnitte am Rande des Zellkerns könnten auch als Schutz gegen externe mechanische Einwirkungen dienen.[7]
Durch die Anhäufung vieler aktiver Gene im Inneren des Zellkerns könnte eine gemeinsame Regulierung dadurch eine Rolle spielen, indem Transkriptionsmaschinerien, Splicingfaktoren usw. geteilt werden. Im Inneren des Zellkerns wurden Transkriptionsfaktoren gefunden, die ihre Gene in direkter Nachbarschaft finden. Dadurch könnte die Expression einfacher reguliert werden und effizienter vor sich gehen.[7]
Mechanismus[Bearbeiten | Quelltext bearbeiten]
Es gibt verschiedene Erklärungen für diesen Mechanismus, der für diese spezielle Anordnung von Chromatin im Zellkern verantwortlich ist. Das biophysikalische Konzept des „Macromolecular crowding“ von Zimmerman und Minton[10] besagt eine Veränderung der molekularen Interaktionen als Antwort auf eine Veränderung der Crowding-Konditionen. „Crowding“ wird hier definiert als das Vorkommen von Makromolekülen, die ein bestimmtes Volumen einnehmen. Sie vermindern damit das verfügbare Volumen für andere Moleküle. Die ursprünglich weit verteilten Moleküle formen kompakte Kompartimente in einem von Molekülen überfüllten System. Eine dichtere Kompaktierung führt zu einem weniger ausgeschlossenen Volumen, in dem sich die Moleküle bewegen können. Dadurch steigt die Entropie des gesamten Systems. Die Bindung von Heterochromatin an die Lamina des Zellkerns könnte ebenso eine Rolle bei der Anordnung von Chromatin im Zellkern spielen. Ist dieser Mechanismus gestört, kann es zu gravierenden Krankheiten kommen.[7]
Aufbau[Bearbeiten | Quelltext bearbeiten]

Die DNA liegt im Zellkern nicht als freies, nacktes Molekül vor, sondern als Chromatin. In einem ersten Schritt wird sie mit Histonen zu Nukleosomen verpackt. Die Bildung der Nukleosomen ergibt eine 6-fache Verkürzung. Varianten von Histonen, die in Nukleosomen vorhanden sein können, posttranslationale Modifikationen und andere Markierungen können die DNA-Zugänglichkeit für die Transkription auf dieser Verpackungsebene steuern. Eine weitere Chromatinkondensation zu 30 nm Fasern (d. h. in Zickzack- oder Solenoid-Form) wurde vorgeschlagen, ist aber nicht sicher. Die Struktur und Organisation von Chromatinschleifen höherer Ordnung innerhalb eines Chromosomenterritoriums ist ebenfalls unklar.[2]
Es gibt Hinweise, dass das Chromatin in Einheiten höherer Ordnung mit jeweils ~1 Mb Länge verpackt ist. Einzelne ∼1-Mb-Domänen könnten aus kleineren Schleifen-Domänen aufgebaut sein, während größere Chromatin-Cluster aus Clustern mehrerer ∼1-Mb-Domänen bestehen könnten. Chromosomenterritorien, die durch eine Fluoreszenz-in-situ-Hybridisierung (3D-FISH) visualisiert werden, erscheinen als Strukturen mit vielfältigen Formen, die aus Chromatin-Domänen höherer Ordnung bestehen.[8]
Die ∼1 Mb-Chromatin-Domänen könnten die grundlegende Struktureinheiten sein, die Chromosomenterritorien aufbauen. Diese Domänen wurden zuerst in S-Phasen-Kernen als Replikationsschwerpunkte dargestellt und erwiesen sich später als dauerhafte Chromatinstrukturen höherer Ordnung. Bislang ist weder ihre ultrastrukturelle Organisation noch die Verpackung von Chromatin, das diese Bereiche verbindet, vollständig geklärt.[8]
Bei Chromosomenterritorien kann man zeigen, dass sie in Areale der frühen DNA-Replikation aufgeteilt sind. Diese haben gendichte 1Mb-Domänen, die im Inneren der Zellkerne liegen. Um sie herum liegen genärmere Abschnitte mit mittlerem bis spätem Replikationszeitpunkt. Diese liegen mehr an der Kernperipherie und haben Kontakt zur Kernlamina. Um den Nukleolus liegen ebenfalls genärmere 1Mb-Domänen mit einem späten Replikationszeitpunkt. Für diese höhere Ordnung innerhalb des Zellkerns könnte eine Hierarchie der Chromatinfibern verantwortlich sein.[1]
Modelle[Bearbeiten | Quelltext bearbeiten]
Mit der Entwicklung von biochemischen Hochdurchsatztechniken wie 3C („Chromosom conformation capture“) und 4C („Chromosom conformation capture-on-chip“ und „circular chromosome conformation capture“) wurden zahlreiche räumliche Wechselwirkungen zwischen benachbarten chromosomalen Abschnitten beschrieben.[2] Diese Beschreibungen wurden ergänzt durch die Erstellung von räumlichen Karten für das gesamte Genom. Zusammen mit den Daten von Fluoreszenz-in-situ-Hybridisierungen haben diese Beobachtungen und physikalische Simulationen zu verschiedenen Modellen der räumlichen Organisation von Chromosomenterritorien geführt.[2]
Sowohl das ICN-Modell als auch die CT-IC-Modelle sind in der Lage, funktionelle Chromatin-Interaktionen zu erklären. Auch können beide Modelle intra- und interchromosomale Umlagerungen erklären. Forscher präferieren die Chromatin-Domänenansicht für die Kernorganisation, so wie es das CT-IC Modell liefert. Dies basiert auf der Überzeugung, dass dieses Modell am besten zu den aktuellen experimentellen Erkenntnissen passt.[11] Obwohl die hier vorgestellten Modelle in einigen Punkten nicht übereinstimmen, schließen sie sich keineswegs gegenseitig aus.[12]
Interchromosome Domain Model (ICD)[Bearbeiten | Quelltext bearbeiten]
Im Interchromosome Domain Modell (ICD) von 1993[13] sind Chromosomenterritorien durch eine glatte Oberfläche von dem sogenannten Interchromatinbereich abgrenzt. Dieser Bereich bildet ein Netzwerk aus verzweigten Kanälen, die sich von den Kernporen über den gesamten Zellkern erstrecken. In diesen Kanälen befinden sich Faktoren für die Transkription, die DNA-Reparatur und für Spleißen der RNA.[7]
Aktive Gene stehen in direktem Kontakt mit dem ICD-Kompartiment, da sie an der Oberfläche von Chromosomenterritorien oder den intrachromosomalen Kanälen liegen. Seltene Chromatinschleifen, die sich von Chromosomenterritorien aus erstrecken, können in den ICD-Raum eindringen, der voraussichtlich wenig oder gar kein Chromatin enthält. Weitere Beobachtungen, dass Transkription auch innerhalb von Chromosomenterritorien auftritt, führten zur Infragestellung dieses Modells, welches die Abläufe zu vereinfacht darstellt.[7]
Chromosome territory – Interchromatin compartment (CT-IC)[Bearbeiten | Quelltext bearbeiten]
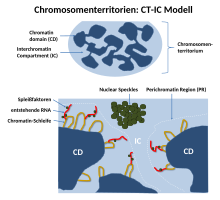
Da beobachtet wurde, dass Transkription nicht nur auf den Rand eines Chromosomenterritoriums beschränkt ist, führten Thomas und Christoph Cremer das „chromosome territory – interchromatin compartment“-Modell (CT-IC-Modell)[14] ein. Sie entwickelten hierfür das ICD-Modell weiter.[7] In diesem Modell werden zwei Bereiche in einem Zellkern beschrieben: Chromosomenterritorien (CTs) und das Interchromatin-Kompartiment (IC). In diesem Modell bauen Chromosomenterritorien ein miteinander verbundenes Chromatin-Netzwerk auf, das mit einem angrenzenden Raum, dem Interchromatin-Kompartiment, verbunden ist. Letzteres kann sowohl mit Hilfe der Licht- als auch der Elektronenmikroskopie beobachtet werden.[2]
Das IC besteht aus einem DNA-freien oder zumindest weitgehend freien, zusammenhängenden Kanalraum, der an den Kernporen beginnt und sich durch größere Kanäle und Lücken zwischen dem zuvor beschriebenen Chromatin-Netzwerk höherer Ordnung erweitert.[8] Das IC beherbergt Nuclear Speckles und eine Vielzahl von weiteren Kernkörperchen. Innerhalb eines einzigen Chromosomenterritoriums wird das Chromosom in definierte Bereiche unterteilt, die auf dem Grad der Chromosomenkondensation basieren.[2] Hier besteht der innere Teil des Interphasen-Chromosoms aus kondensierteren Chromatin-Domänen oder Chromatinfasern höherer Ordnung. Diese bestehen aus mehreren ~1 Mb großen DNA-Domänen, die den Replikationszentren entsprechen.[7] Diese sogenannten 1-Mb-Domänen sollen wiederum aus vielen kleinen 30-200 kb enthaltenden DNA-Schleifen bestehen.[7] Zwischen diesen 1-Mb-Domänen und dem Interchromatin-Kompartiment befindet sich eine dünne (<200 nm) Schicht aus dekondensiertem Chromatin, die Perichromatin-Region (PR) genannt wurde. Auch die Chromosomenterritorien selbst werden hier durch die Kanäle des Interchromatin-Kompartiments durchzogen. Wie anhand elektronenmikroskopischer Aufnahmen gezeigt werden konnte, bestehen die Perichromatin-Regionen aus dekondensierten Chromatin-Schleifen. Dadurch bilden sie eine Schwamm-ähnliche Struktur und sind somit zugänglich für die Proteine des Interchromatin-Kompartiment.[2][7]
Funktionell stellt die Perichromatin-Region das wichtigste Transkriptionskompartiment dar. Dies ist auch die Region, in der das RNA-Spleißen stattfindet. Die DNA-Replikation und die DNA-Reparatur wird ebenfalls überwiegend innerhalb der Perichromatin-Region durchgeführt. Schließlich werden auch im Perichromatin-Bereich generierte RNA-Transkripte, so genannte Perichromatin-Fibrillen, erzeugt.[2] Perichromatin-Fibrillen werden dann durch die Enzyme aus dem Interchromatin-Kompartiment bei Bedarf gespleißt.[2][8] Die PR ist mit regulatorischen und kodierenden Sequenzen aktiver Gene angereichert und stellt die wichtigste (jedoch nicht ausschließliche) nukleäre Unterabteilung für Transkription, RNA-Spleißen, DNA-Replikation und Reparatur dar.[15] Stan Fakan et al. zeigten um das Jahr 2000, dass Transkription und DNA-Replikation vorzugsweise innerhalb der PR durchgeführt werden.[15][16]
In dem CT-IC Modell sollten sich aktive Gene mehr an der Oberfläche der gefalteten Chromatinfasern, inaktive mehr im Inneren der Chromatinstruktur befinden. Um auch innenliegende Gene der Transkription zugänglich zu machen, würde bei Bedarf die Chromatinstruktur aufgelockert.[1] Kontinuierliche, eingeschränkte Chromatinbewegungen wurden von der Ebene einzelner ∼1 Mbp-Chromatindomänen bis hin zu ganzen CTs beobachtet. Solche Bewegungen erzwingen kontinuierliche Änderungen der tatsächlichen Breite von IC-Kanälen und Lakunen (Hohlräume, engl. „lacunas“). Dies bietet Möglichkeiten für dynamische Änderungen der Chromatin-Interaktionen.[17] Nach dem CT-IC-Modell weisen Chromosomenterritorien und auch Chromatin-Domänen (z. B. p- und q-Arme) eine geringe oder gar keine Vermischung (Intermingling) zwischen ihnen auf, wie einige Studien zeigen. Obwohl das CT-IC-Modell eine robuste und gut akzeptierte Hypothese ist, muss noch betätigt werden, ob es Strukturen höherer Ordnung wie die 100 kbp-Domäne gibt und welche Funktionselemente eine solche Organisation antreiben.[12]
CT-IC ANC-INC[Bearbeiten | Quelltext bearbeiten]
Das CT-IC ANC-INC Modell erweitert das CT-IC Modell. Es integriert aktuelle Erkenntnisse über die strukturelle Organisation des Zellkerns in einen Rahmen aus zwei strukturell und funktionell miteinander verflochtenen Kompartimenten, das aktive Kernkompartiment (Active nuclear compartment – ANC) und das inaktive Kernkompartiment (Inactive nuclear compartment – INC).[15][18]
Nach diesem Modell wird ein Chromosomenterritorium aus „topologisch assoziierten Chromatindomänen“ (TAD) aufgebaut. Diese bilden ein Netzwerk von Chromatin-Domain-Clustern (CDCs).[18]
Das aktive Kernkompartiment ANC beinhaltet DNA mit einer niedrigen Dichte. Es enthält aktive Gene, die aktuell abgelesen werden. Es ist angereichert mit epigenetischen Markierungen für transkriptionsfähiges Chromatin und RNA-Polymerase II. Das ANC ist der wichtigste Ort der RNA-Synthese. Es enthält zwei wesentliche strukturelle Einheiten, das Interchromatin-Kompartiment (IC) und die Perichromatin-Region (PR). Die Perichromatin-Region (PR) wird durch die transkriptionell aktive Peripherie der Chromatin-Domänen-Clustern (CDCs) des ANC gebildet. Diese Bereiche sind weniger verdichtet. Regulatorische Sequenzen von Genen können innerhalb des ANC exponiert werden, unabhängig davon, ob Gene aktiv oder inaktiv sind.[15][17] Chromatin-Schleifen können aus den CDCs/TADs herausragen und als Hauptorte der RNA-Synthese dienen.[18]
Das inaktive Kernkompartiment (INC) hat eine hohe DNA-Dichte, welches die in dieser Zelle stillgelegten Gene enthält. Das INC umfasst den kompakten, transkriptionell inaktiven Kern von CDCs.[15][17] In der konventionellen Terminologie kann das INC als Teil des fakultativen Heterochromatins betrachtet werden.[15]
In welcher Weise das ANC und das INC zusammen interagieren ist noch Gegenstand aktueller Forschung. So wird das Szenario überlegt, dass nur regulatorische Sequenzen, die in einer bestimmten Zelle aktiv verwendet werden, innerhalb des ANC lokalisiert sind, während solche Sequenzen von Genen, die in dieser Zelle dauerhaft abgeschaltet sind, in das INC zurückgezogen werden, das durch das kompakte und weitgehend unzugängliche Innere der CDCs repräsentiert wird. Oder regulatorische Sequenzen von Genen sind immer innerhalb des ANC zugänglich, unabhängig davon, ob Gene aktiv oder inaktiv sind.[17]
Das IC steht in Verbindung mit den Kernporen und dient dem nukleären Import und Export von Molekülen.[17] IC-Kanäle ermöglichen z. B. eine vereinfachte Bewegung von mRNPs entlang der Kanäle zu den Kernporen. Sie bieten bevorzugte Wege für funktionelle Proteine, wie z. B. Transkriptionsfaktoren (TFs), die in das Innere des Kerns eindringen und zu ihren DNA-Bindungsorten führen. Und sie ermöglichen eine schnelle intranukleäre Verteilung von Faktoren, die in Kernkörperchen gespeichert sind, sowie von regulatorischen, nicht-kodierenden RNAs von Stellen, an denen solche RNAs synthetisiert werden, zu Stellen, an denen solche Faktoren und RNAs benötigt werden.[15]
ICN[Bearbeiten | Quelltext bearbeiten]
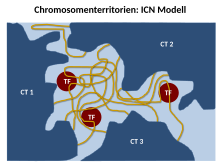
Das ICN-Modell (Interchromatin Network) von Branco und Pombo[19] sagt voraus, dass vermischte Chromatinfasern/Schleifen sowohl cis- (innerhalb desselben Chromosoms) als auch trans- (zwischen verschiedenen Chromosomen) Kontakte herstellen können. Diese Vermischung/Verflechtung (Intermingling) ist einheitlich. Deshalb ist die Unterscheidung zwischen Chromosomenterritorium und Interchromatin-Kompartiment funktionell bedeutungslos.[2] Anhand von FISH-Experimenten auf ultradünnen Gefrierschnitten konnten sie zeigen, dass bis zu 20–30% des Volumens eines Chromosomenterritoriums mit dem Nachbarterritorium geteilt werden kann.[7][12] Simulationen der Chromosomentranslokationen, die auf Modellen der Chromosomenorganisation basieren, haben auf die Existenz eines signifikanten Maßes an Vermischung von DNA zwischen CTs hingewiesen.[19]
Das ICN-Modell schlägt vor, dass das Gleichgewicht zwischen gewebespezifischen intra- und interchromosomalen Assoziationen die Position und Konformation des Chromosoms definieren wird. Dies geschieht zusammen mit einer funktionellen Bindung an andere nukleäre Landmarken wie der Lamina und den Nukleoli.[12] In diesem Interchromatin-Netzwerk können sich Schleifen von einer CT ausdehnen, um Schleifen von einem anderen CT zu treffen.[8] Schleifenbildung konnte erstmals in Säugetierzellen nachgewiesen werden, wo eine Kontrollregion physikalisch mit aktiven Genen interagiert, die 50 kb entfernt liegen.[12]
Die Verflechtung konnte auf der ultrastrukturellen Ebene bestätigt werden, nachdem man die Chromosomen mit der Immungoldfärbung markiert und mit einem Elektronenmikroskop abgebildet hatte. Es besteht daher ein hohes Potenzial für interchromosomale Wechselwirkungen, was sich in der Häufigkeit der Chromosomenumlagerungen widerspiegelt. Tatsächlich korreliert das Ausmaß der Vermischung zwischen Paaren von CTs mit ihrem Translokationspotenzial.[12]
Der Vorteil des ICN-Modells besteht darin, dass es eine hohe Chromatindynamik und diffusionsähnliche Bewegungen ermöglicht. Die Autoren schlagen vor, dass die fortlaufende Transkription den Grad der Vermischung zwischen spezifischen Chromosomen beeinflusst, indem sie die Assoziationen zwischen bestimmten Orten stabilisiert. Solche Wechselwirkungen hängen wahrscheinlich von der transkriptionellen Aktivität der Loci ab und sind daher zelltypspezifisch.[2]
Lattice-Modell[Bearbeiten | Quelltext bearbeiten]
Das von Dehgani et al. vorgeschlagene Lattice-Modell (Gittermodell) basiert auf der Erkenntnis, dass die Transkription auch innerhalb der inneren, stärker verdichteten Chromosomenterritorien stattfindet und nicht nur an der Schnittstelle zwischen dem Interchromatin-Kompartiment und der Perichromatin-Region. Unter Verwendung von ESI (electron spectroscopic imaging) zeigten Dehgani, dass Chromatin als eine Anordnung von Desoxy-Ribonukleoproteinfasern mit einem Durchmesser von 10–30 nm organisiert war. In der Studie von Deghani waren die Interchromatin-Kompartimente, die im CT-IC-Modell als große Kanäle zwischen Chromosomenterritorien beschrieben werden, nicht sichtbar. Stattdessen schufen Chromatinfasern ein loses Geflecht aus Chromatin im gesamten Kern, das sich an der Peripherie von Chromosomenterritorien vermischte. So sind die inter- und intra-chromosomalen Räume innerhalb dieses Netzwerks im Wesentlichen zusammenhängend und bilden zusammen den intra-nukleären Raum.[2]
Weitere Modelle[Bearbeiten | Quelltext bearbeiten]
Das Fraser and Bickmore Modell betont die funktionelle Bedeutung von riesigen Chromatinschleifen, die aus Chromosomenterritorien stammen und sich über den nukleären Raum erstrecken, um Transkriptionsfabriken zu teilen. In diesem Fall können sowohl cis- als auch trans-Schleifen von dekondensiertem Chromatin von derselben Transkriptionsfabrik co-exprimiert und co-reguliert werden.[2]
Die Chromatin-Polymer-Modelle gehen von einem breiten Spektrum von Chromatinschleifengrößen aus. Sie prognostizieren die beobachteten Abstände zwischen genomischen Loci und Chromosomenterritorien sowie die Wahrscheinlichkeit, dass Kontakte zwischen bestimmten Loci gebildet werden. Diese Modelle wenden physikalische Ansätze an, die die Bedeutung der Entropie für das Verständnis der Kernorganisation verdeutlichen.[2]
Literatur[Bearbeiten | Quelltext bearbeiten]
- Rieke Kempfer, Ane Pombo: Methods for mapping 3D chromosome architecture. In: Nat Rev Genet 21, 4, 2020: 207–226. doi: 10.1038/s41576-019-0195-2.
- Andrew J Fritz, Nitasha Sehgal, Artem Pliss, Jinhui Xu, Ronald Berezney: Chromosome territories and the global regulation of the genome. In: Genes Chromosomes Cancer 58, 7, 2019: 407–426. PMC 7032563 (freier Volltext)
- Leah F Rosin, Olivia Crocker, Randi L Isenhart, Son C Nguyen, Zhuxuan Xu, Eric F Joyce: Chromosome territory formation attenuates the translocation potential of cells. In: eLife 8, 2019: e49553. PMC 6855801 (freier Volltext)
- Teresa Szczepińska, Anna Maria Rusek, Dariusz Plewczynski: Intermingling of chromosome territories. In: Genes Chromosomes Cancer 58, 7, 2019: 500–506. doi:10.1002/gcc.22736
- Thomas Cremer, Marion Cremer: Chromosome territories. In: Cold Spring Harb Perspect Biol 2, 3, 2010: a003889. PMC 2829961 (freier Volltext) ← Zahlreiche Bilder.
Quellenangaben[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c d e Robert Mayer: Anordnung und Struktur von Chromosomenterritorien in Mauszellen: Zelltypspezifische Unterschiede und Gemeinsamkeiten. (PDF) 1. Februar 2006, abgerufen am 7. Juli 2019 (Dissertation an der Fakultät für Biologie Ludwig-Maximilians-Universität München).
- ↑ a b c d e f g h i j k l m n What are chromosomes and chromosome territories? Abgerufen am 7. Juli 2019 (englisch).
- ↑ C. Zorn, Thomas Cremer, Christoph Cremer, Jürgen Zimmer: Laser UV microirradiation of interphase nuclei and post-treatment with caffeine. A new approach to establish the arrangement of interphase chromosomes. In: Hum Genet 35, 1, 1976: 83–89. PMID 1002167
- ↑ Margit Schardin, Thomas Cremer, H D Hager, M Lang: Specific staining of human chromosomes in Chinese hamster x man hybrid cell lines demonstrates interphase chromosome territories. In: Hum Genet 71, 4, 1985: 281–287. PMID 2416668
- ↑ Laura Manuelidis: Individual interphase chromosome domains revealed by in situ hybridization. In: Hum Genet 71, 4, 1985, 288–293. PMID 3908288
- ↑ Martin A. Lysak, Ales Pecinka, Ingo Schubert: Recent progress in chromosome painting of Arabidopsis and related species. In: Chromosome Res 11, 3, 2003: 195–204. doi:10.1023/A:1022879608152.
- ↑ a b c d e f g h i j k l m Kathrin Teller: Die Architektur des X-Chromosoms – eine „Top down“-Analyse in humanen Fibroblasten. (PDF) 28. Februar 2008, abgerufen am 7. Juli 2019 (Dissertation der Fakultät für Biologie der Ludwig-Maximilians-Universität München).
- ↑ a b c d e f g T. Cremer, M. Cremer: Chromosome Territories. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 3, 1. März 2010, ISSN 1943-0264, S. a003889–a003889, doi:10.1101/cshperspect.a003889 (cshlp.org [abgerufen am 7. Juli 2019]).
- ↑ T. C. Hsu, Kurt Benirschke: Erignathus barbatus (Bearded seal). In: An Atlas of Mammalian Chromosomes. Springer New York, New York, NY 1975, ISBN 978-1-4684-7996-6, S. 137–140, doi:10.1007/978-1-4615-6434-8_35 (springer.com [abgerufen am 7. Juli 2019]).
- ↑ Steven B Zimmerman, Allen P Minton: Macromolecular Crowding: Biochemical, Biophysical, and Physiological Consequences. In: Annual Review of Biophysics and Biomolecular Structure. Band 22, Nr. 1, Juni 1993, ISSN 1056-8700, S. 27–65, doi:10.1146/annurev.bb.22.060193.000331.
- ↑ Robert A. Meyers: Epigenetic Regulation and Epigenomics. Hrsg.: Wiley-Blackwell. ISBN 978-3-527-32682-2, S. 453–473.
- ↑ a b c d e f Miguel R. Branco, Ana Pombo: Chromosome organization: new facts, new models. In: Trends in Cell Biology. Band 17, Nr. 3, März 2007, S. 127–134, doi:10.1016/j.tcb.2006.12.006 (elsevier.com [abgerufen am 7. Juli 2019]).
- ↑ T. Cremer, A. Kurz, R. Zirbel, S. Dietzel, B. Rinke: Role of Chromosome Territories in the Functional Compartmentalization of the Cell Nucleus. In: Cold Spring Harbor Symposia on Quantitative Biology. Band 58, Nr. 0, 1. Januar 1993, ISSN 0091-7451, S. 777–792, doi:10.1101/SQB.1993.058.01.085 (cshlp.org [abgerufen am 26. Juni 2019]).
- ↑ T. Cremer, C. Cremer: Chromosome territories, nuclear architecture and gene regulation in mammalian cells. In: Nature Reviews Genetics. Band 2, Nr. 4, April 2001, ISSN 1471-0056, S. 292–301, doi:10.1038/35066075.
- ↑ a b c d e f g T. Cremer, M. Cremer, C. Cremer: The 4D Nucleome: Genome Compartmentalization in an Evolutionary Context. In: Biochemistry (Moscow). Band 83, Nr. 4, April 2018, ISSN 0006-2979, S. 313–325, doi:10.1134/S000629791804003X.
- ↑ Françoise Jaunin, Astrid E. Visser, Dusan Cmarko, Jacob A. Aten, Stanislav Fakan: Fine Structural in Situ Analysis of Nascent DNA Movement Following DNA Replication. In: Experimental Cell Research. Band 260, Nr. 2, November 2000, S. 313–323, doi:10.1006/excr.2000.4999 (elsevier.com [abgerufen am 7. Juli 2019]).
- ↑ a b c d e Thomas Cremer, Marion Cremer, Barbara Hübner, Hilmar Strickfaden, Daniel Smeets: The 4D nucleome: Evidence for a dynamic nuclear landscape based on co-aligned active and inactive nuclear compartments. In: FEBS Letters. Band 589, 20PartA, 7. Oktober 2015, S. 2931–2943, doi:10.1016/j.febslet.2015.05.037 (wiley.com [abgerufen am 7. Juli 2019]).
- ↑ a b c Christoph Cremer: Mobilität und Dynamik im Zellkern. In: Heidelberger Jahrbücher Online. 10. September 2018, S. Band 3 (2018): Perspektiven der Mobilität–, doi:10.17885/heiup.hdjbo.2018.0.23823 (uni-heidelberg.de [abgerufen am 7. Juli 2019]).
- ↑ a b Miguel R Branco, Ana Pombo: Intermingling of Chromosome Territories in Interphase Suggests Role in Translocations and Transcription-Dependent Associations. In: PLoS Biology. Band 4, Nr. 5, 25. April 2006, ISSN 1545-7885, S. e138, doi:10.1371/journal.pbio.0040138 (plos.org [abgerufen am 26. Juni 2019]).