Sentido (biología molecular)

En biología molecular y genética, el sentido de las moléculas de ácido nucleico (a menudo ADN o ARN) es la naturaleza de sus funciones y las funciones de sus unidades de ácido nucleico de las moléculas complementarias en la especificación de aminoácidos. Dependiendo del contexto dentro de la biología molecular, el sentido puede tener significados ligeramente diferentes.
Sentido del ADN
[editar]Los biólogos moleculares llaman a una sola cadena de sentido del ADN (o positivo (+)) si una versión de ARN de la misma secuencia se traduce en proteína. Su cadena complementaria se llama antisentido (o sentido negativo (-) ). A veces se encuentran las frases que codifican cadenas (sentido) y plantilla de cadena (antisentido); sin embargo, los ARN codificantes y no codificantes de proteínas pueden transcribirse a partir de la cadena sentido. Además, los términos "sentido" y "antisentido" son relativos a la transcripción de ARN en cuestión y no a la cadena de ADN en su conjunto. En otras palabras, cualquiera de las cadenas de ADN puede servir como cadena sentido o antisentido para una transcripción de ARN particular. En algunos casos, las transcripciones de ARN pueden transcribirse en ambas direcciones (es decir, en cualquiera de las cadenas) desde una región promotora común, o pueden transcribirse desde dentro de los intrones en cualquiera de las cadenas.[1][2][3]
ADN antisentido
[editar]Las dos cadenas complementarias de ADN bicatenario (ADNds) generalmente se diferencian como la cadena "sentido" y la cadena "antisentido". La cadena de sentido del ADN se parece al ARN mensajero (ARNm) y puede usarse para leer el código de proteína esperado; por ejemplo, ATG en el sentido de ADN puede corresponder a un codón AUG en el ARNm, que codifica el aminoácido metionina. Sin embargo, la cadena de sentido del ADN en sí misma no se usa para producir proteínas en la célula. Es la cadena antisentido de ADN que sirve como fuente del código de la proteína, porque, con bases complementarias a la cadena de sentido del ADN, se usa como plantilla para el ARNm. Dado que la transcripción da como resultado un producto de ARN complementario a la cadena molde de ADN, el ARNm es complementario a la cadena antisentido de ADN. El ARNm es lo que se usa para la traducción (síntesis de proteínas).
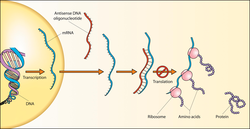
Por lo tanto, un triplete de base 3'-TAC-5 'en la cadena antisentido de ADN se puede usar como plantilla que dará como resultado un triplete de base 5'-AUG-3' en ARNm (AUG es el codón para la metionina, el codón de inicio). La cadena de sentido del ADN tendrá el triplete ATG, que se parece a AUG pero no se usará para producir metionina porque no se usará para producir ARNm. La cadena de sentido del ADN se llama cadena de "sentido" no porque se usará para producir proteínas (no lo será), sino porque tiene una secuencia que se parece a la secuencia del codón de la proteína.
En biología e investigación, las moléculas antisentido cortas pueden interactuar con cadenas complementarias de ácidos nucleicos, modificando la expresión de genes.
Ejemplo con ADN bicatenario
[editar]Cadena de ADN 1: cadena antisentido (transcrita a) → cadena de ARN (sentido)
Cadena de ADN 2: cadena sensorial
Algunas regiones dentro de una doble cadena de ADN codifican genes, que generalmente son instrucciones que especifican el orden de los aminoácidos en una proteína junto con secuencias reguladoras, sitios de empalme, intrones no codificantes y otros detalles complicados. Para que una célula use esta información, una cadena del ADN sirve como plantilla para la síntesis de una cadena complementaria de ARN. La cadena de ADN de plantilla se llama cadena transcrita con secuencia antisentido y se dice que el transcrito de ARNm es secuencia de sentido (el complemento de antisentido). Debido a que el ADN es bicatenario, la cadena complementaria a la secuencia antisentido se llama cadena no transcrita y tiene la misma secuencia de sentido que el transcrito de ARNm (aunque las bases T en el ADN están sustituidas con bases U en el ARN).
| 3'CGCTATAGCGTTT 5' | Cadena antisentido de ADN (plantilla/no codificación) | Utilizado como plantilla para la transcripción. |
| 5'GCGATATCGCAAA 3' | Cadena de sentido del ADN (sin plantilla/codificación) | Complementario a la sección de la plantilla. |
| 5'GCGAUAUCGCAAA 3' | transcripción de sentido de ARNm | Cadena de ARN que se transcribe de la cadena no codificante (plantilla / antisentido). Nota1 : Excepto por el hecho de que todas las timinas son ahora uracilos (T -> U), es complementario a la cadena de ADN no codificante (plantilla / antisentido) ( idéntica a la cadena de ADN codificante (no plantilla / sentido)). |
| 3'CGCUAUAGCGUUU 5' | transcripción antisentido de ARNm | Cadena de ARN que se transcribe de la cadena de codificación (no plantilla / sentido). Nota: Excepto por el hecho de que todas las timinas son ahora uracilos (T -> U), es complementario a la cadena de ADN codificante (no plantilla / sentido) ( idéntica a la cadena de ADN no codificante (plantilla / antisentido)). |
Una nota sobre la confusión entre las cadenas de "sentido" y "antisentido": los nombres de las cadenas realmente dependen de la dirección en la que está escribiendo la secuencia que contiene la información para las proteínas (la información de "detección"), no en qué cadena está en la parte superior o inferior (que es arbitrario). La única información biológica real que es importante para marcar hebras es la ubicación del grupo fosfato 5 'y el grupo hidroxilo 3' porque estos extremos determinan la dirección de transcripción y traducción. Una secuencia 5 'CGCTAT 3' es equivalente a una secuencia escrita 3 'TATCGC 5' siempre que se anoten los extremos 5 'y 3'. Si los extremos no están etiquetados, la convención es suponer que la secuencia se escribe de izquierda a derecha en la dirección de 5 'a 3'. La hebra de Watson se refiere a la hebra superior de 5 'a 3' (5 '→ 3'), mientras que la hebra Crick se refiere a la hebra inferior de 5 'a 3' (3 '← 5').[4] Las cadenas de Watson y Crick pueden ser cadenas sentido o antisentido dependiendo del gen cuyas secuencias se muestran en la base de datos de secuencias del genoma. Por ejemplo, YEL021W, un alias del gen URA3 utilizado en la base de datos de NCBI, define que este gen se encuentra en el 21 ° marco de lectura abierto (ORF) del centrómero del brazo izquierdo (L) del número de cromosoma V (E) de la levadura (Y) ), y que la cadena de codificación de expresión es la cadena de Watson (W). YKL074C define el 74.º ORF a la izquierda del centrómero del cromosoma XI y denota la cadena de codificación de la cadena de Crick (C). Otro término confuso que se refiere al filamento "más" y "menos" también se usa ampliamente. Si la cadena es sentido (positivo) o antisentido (negativo), la secuencia de consulta predeterminada en la alineación de NCBI BLAST es la cadena "Plus".
Ambisentido
[editar]Se dice que un genoma monocatenario que contiene sentido positivo y sentido negativo es ambisentido. Los bunyavirus tienen 3 fragmentos de ARN monocatenario (ARNss), algunos de los cuales contienen secciones con sentido positivo y negativo; los arenavirus también son virus ARNss con un genoma ambisentido, ya que tienen 2 fragmentos que son principalmente de sentido negativo, excepto por parte de los extremos 5 'de los segmentos grandes y pequeños de su genoma.
ARN antisentido
[editar]El ARN antisentido es un transcrito de ARN que es complementario al ARNm endógeno. En otras palabras, es una cadena no codificante complementaria a la secuencia codificante de ARN; Esto es similar al ARN viral de sentido negativo. La introducción de una codificación transgénica para el ARN antisentido es una técnica utilizada para bloquear la expresión de un gen de interés. El ARN antisentido marcado radiactivamente puede usarse para mostrar el nivel de transcripción de genes en varios tipos de células. Algunos tipos estructurales antisentido alternativos se están aplicando experimentalmente como terapia antisentido, con al menos una terapia antisentido aprobada para su uso en humanos.
Cuando el ARNm forma un dúplex con una secuencia de ARN antisentido complementaria, la traducción se bloquea. Este proceso está relacionado con la interferencia de ARN.
Las moléculas de ácido nucleico antisentido se han utilizado experimentalmente para unirse al ARNm y evitar la expresión de genes específicos. Las terapias antisentido también están en desarrollo; en los EE. UU., la Administración de Alimentos y Medicamentos (FDA) ha aprobado los oligos fomivirsen (Vitravene) y mipomersen (Kynamro)[5] antisentido de fosforotioato para uso terapéutico en humanos.
Las células pueden producir moléculas de ARN antisentido de forma natural, llamadas microARN, que interactúan con moléculas de ARNm complementarias e inhiben su expresión.
Sentido de ARN en virus
[editar]En virología, se puede decir que el genoma de un virus de ARN tiene sentido positivo, también conocido como "cadena positiva" o sentido negativo, también conocido como "cadena negativa". En la mayoría de casos, los términos sentido y la hebra se usan indistintamente, haciendo tales términos como equivalentes de cadena positiva de de sentido positivo, y de la cadena positiva equivalente a de sentido positivo. Si un genoma de virus tiene sentido positivo o sentido negativo puede usarse como base para clasificar los virus.
Sentido positivo
[editar]El ARN viral de sentido positivo (5 'a 3') significa que una secuencia particular de ARN viral puede traducirse directamente en las proteínas virales deseadas. Por lo tanto, en los virus de ARN de sentido positivo, el genoma de ARN viral puede considerarse ARNm viral y la célula huésped puede traducirlo inmediatamente. A diferencia del ARN de sentido negativo, el ARN de sentido positivo tiene el mismo sentido que el ARNm. Algunos virus (p. Ej., Coronaviridae) tienen genomas de sentido positivo que pueden actuar como ARNm y usarse directamente para sintetizar proteínas sin la ayuda de un intermediario de ARN complementario. Debido a esto, estos virus no necesitan tener una ARN polimerasa empaquetada en el virión.
Sentido negativo
[editar]El ARN viral de sentido negativo (3 'a 5') es complementario al ARNm viral, por lo tanto, un ARN polimerasa dependiente de ARN debe ser producido a partir de él antes de la traducción. El ARN de sentido negativo (como el ADN) tiene una secuencia de nucleótidos complementaria al ARNm que codifica. Al igual que el ADN, este ARN no puede traducirse directamente en proteínas . En cambio, primero debe transcribirse en un ARN de sentido positivo que actúa como un ARNm. Algunos virus (Influenza, por ejemplo) tienen genomas de sentido negativo y, por lo tanto, deben llevar una ARN polimerasa dentro del virión .
Oligonucleótidos antisentido
[editar]El silenciamiento génico se puede lograr mediante la introducción en las células de un "oligonucleótido antisentido" corto que es complementario a una diana de ARN. Este experimento fue realizado por primera vez por Zamecnik y Stephenson en 1978[6] y continúa siendo un enfoque útil, tanto para experimentos de laboratorio como para aplicaciones clínicas (terapia antisentido).[7] Varios virus como los virus de la gripe,[8][9][10][11] El virus sincitial respiratorio (RSV) y el coronavirus del SARS (SARS-CoV) se han dirigido utilizando oligonucleótidos antisentido para inhibir su replicación en células huésped.
Si el oligonucleótido antisentido contiene un tramo de ADN o un imitador de ADN (ADN de fosforotioato, 2'F-ANA u otros), puede reclutar Ribonucleasa H para degradar el ARN objetivo. Esto hace que el mecanismo de silenciamiento génico sea catalítico. El ARN bicatenario también puede actuar como un agente antisentido catalítico dependiente de enzimas a través de la ruta ARNi/ARNsi, lo que implica el reconocimiento del ARNm objetivo a través del emparejamiento de la cadena antisentido sensorial seguido de la degradación del ARNm objetivo por el complejo silenciador inducido por ARN (RISC). El sistema de plásmido hok/sok R1 proporciona otro ejemplo más de un proceso de regulación antisentido dependiente de enzimas a través de la degradación enzimática del dúplex de ARN resultante.
Otros mecanismos antisentido no dependen de las enzimas, pero implican un bloqueo estérico de su ARN objetivo (por ejemplo, para evitar la traducción o inducir un empalme alternativo). Los mecanismos antisentido de bloqueo estérico a menudo usan oligonucleótidos que están muy modificados. Como no hay necesidad de reconocimiento de Ribonucleasa H, esto puede incluir productos químicos tales como 2'-O-alquilo, ácido nucleico peptídico (PNA), ácido nucleico bloqueado (LNA) y oligómeros de morfolino.
Véase también
[editar]- Terapia antisentido
- Direccionalidad (biología molecular)
- ADN
- Tabla de codones de ADN
- Virus ARN
- Transcripción
- Traducción
- Replica viral
Referencias
[editar]- ↑ Anne-Lise Haenni (2003). «Expression strategies of ambisense viruses». Virus Research 93 (2): 141-150. PMID 12782362. doi:10.1016/S0168-1702(03)00094-7.
- ↑ Kakutani T; Hayano Y; Hayashi T; Minobe Y. (1991). «Ambisense segment 3 of rice stripe virus: the first instance of a virus containing two ambisense segments». J Gen Virol 72 (2): 465-8. PMID 1993885. doi:10.1099/0022-1317-72-2-465.
- ↑ Zhu Y; Hayakawa T; Toriyama S; Takahashi M. (1991). «Complete nucleotide sequence of RNA 3 of rice stripe virus: an ambisense coding strategy». J Gen Virol 72 (4): 763-7. PMID 2016591. doi:10.1099/0022-1317-72-4-763.
- ↑ Cartwright, Reed; Dan Graur (8 de febrero de 2011). «The multiple personalities of Watson and Crick strands». Biology Direct 6: 7. PMC 3055211. PMID 21303550. doi:10.1186/1745-6150-6-7.
- ↑ Staff (29 January 2013) FDA approves new orphan drug Kynamro to treat inherited cholesterol disorder U.S. Food and Drug Administration, Retrieved 31 January 2013
- ↑ Zamecnik, P.C.; Stephenson, M.L. (1978). «Inhibition of Rous sarcoma Virus Replication and Cell Transformation by a Specific Oligodeoxynucleotide». Proc. Natl. Acad. Sci. USA 75 (1): 280-284. PMC 411230. PMID 75545. doi:10.1073/pnas.75.1.280.
- ↑ Watts, J.K.; Corey, D.R. (2012). «Silencing Disease Genes in the Laboratory and in the Clinic». J. Pathol. 226 (2): 365-379. PMC 3916955. PMID 22069063. doi:10.1002/path.2993.
- ↑ Kumar, Binod; Khanna, Madhu; Meseko, Clement A.; Sanicas, Melvin; Kumar, Prashant; Asha, Kumari; Asha, Kumari; Kumar, Prashant et al. (January 2019). «Advancements in Nucleic Acid Based Therapeutics against Respiratory Viral Infections». Journal of Clinical Medicine (en inglés) 8 (1): 6. PMC 6351902. PMID 30577479. doi:10.3390/jcm8010006.
- ↑ Kumar, Binod; Asha, Kumari; Khanna, Madhu; Ronsard, Larance; Meseko, Clement Adebajo; Sanicas, Melvin (10 de enero de 2018). «The emerging influenza virus threat: status and new prospects for its therapy and control». Archives of Virology 163 (4): 831-844. ISSN 0304-8608. PMID 29322273. doi:10.1007/s00705-018-3708-y.
- ↑ Kumar, Prashant; Kumar, Binod; Rajput, Roopali; Saxena, Latika; Banerjea, Akhil C.; Khanna, Madhu (2 de junio de 2013). «Cross-Protective Effect of Antisense Oligonucleotide Developed Against the Common 3′ NCR of Influenza A Virus Genome». Molecular Biotechnology 55 (3): 203-211. ISSN 1073-6085. PMID 23729285. doi:10.1007/s12033-013-9670-8.
- ↑ Kumar, B.; Khanna, Madhu; Kumar, P.; Sood, V.; Vyas, R.; Banerjea, A. C. (9 de julio de 2011). «Nucleic Acid-Mediated Cleavage of M1 Gene of Influenza A Virus Is Significantly Augmented by Antisense Molecules Targeted to Hybridize Close to the Cleavage Site». Molecular Biotechnology 51 (1): 27-36. ISSN 1073-6085. PMID 21744034. doi:10.1007/s12033-011-9437-z.