Negarnaviricota

| Negarnaviricota | ||||||
|---|---|---|---|---|---|---|
 Микрофотографии некоторых вирусов из типа Negarnaviricota, полученные с помощью просвечивающего электронного микроскопа. Не в масштабе. Слева направо, сверху вниз: эболавирус Заир, вирус Син Номбре, респираторно-синцитиальный вирус человека, вирус Хендра, неидентифицированные рабдовирусы, вирус кори | ||||||
| Научная классификация | ||||||
| Международное научное название | ||||||
| Negarnaviricota | ||||||
| Подтипы | ||||||
| Группа по Балтимору | ||||||
| V: (-)оцРНК-вирусы | ||||||
| ||||||
Negarnaviricota (лат.) — тип вирусов из царства Orthornavirae реалма Riboviria[2], имеющих геном, состоящий из негативной (антисмысловой) одноцепочечной рибонуклеиновой кислоты. Относится к V группе классификации вирусов по Балтимору[3], составляя в ней подавляющее большинство видов.
Геном Negarnaviricota представляет комплиментарную цепь, на матрице которой при помощи вирусного фермента РНК-зависимая РНК-полимераза (RdRp) синтезируется матричная РНК (мРНК). Во время репликации генома вируса RdRp синтезирует смысловую (положительную) цепь вирусного РНК-генома, которая используется в качестве матрицы для создания негативных цепей РНК, являющихся новыми копиями вируса. Представители Negarnaviricota обладают рядом других общих характеристик: большинство имеет вирусную оболочку, окружающую капсид с РНК вируса, геном вируса обычно представляет линейную молекулу и он зачастую сегментирован.
Наиболее известные[источник не указан 1196 дней] оц(-)РНК-вирусы, переносимые членистоногими, включают вирус лихорадки Рифт-Валли и вирус пятнистого увядания томатов[англ.]. Примечательные[источник не указан 1196 дней] оц(-)РНК-вирусы позвоночных включают вирус Эбола, Orthohantavirus, вирусы гриппа, вирус лихорадки Ласса и вирус бешенства.
Этимология названия
[править | править код]Название Negarnaviricota состоит из трёх частей: лат. Nega — «отрицательный», rna обозначает РНК, а -viricota является суффиксом, используемым для обозначения ранга тип в номенклатуре вирусов. Название подтипа Haploviricotina состоит из Haplo, от др.-греч. ἁπλός — простой, и суффикса -viricotina, применяемого для обозначения подтипа вирусов. Название Polyploviricotina следует той же схеме: Polyplo от др.-греч. πολύπλοκος — «сложный» и описанный ранее суффикс[3].
Свойства
[править | править код]Геном
[править | править код]
Все вирусы в типе Negarnaviricota являются одноцепочечными РНК-вирусами с негативной цепью. Последнее означает, что информационная РНК (мРНК) синтезируется с геномной РНК вирусным ферментом РНК-зависимой РНК-полимеразой (RdRp), также называемый РНК-репликазой, который кодируется всеми оц(-)РНК-вирусами. За исключением вирусов рода Tenuivirus и некоторых вирусов семейства Chuviridae, все оц(-)РНК-вирусы имеют линейный, а не кольцевой геном и эти геномы могут состоять из одного или нескольких сегментов[3][4][5]. Все оц(-)-РНК-вирусы содержат концевые инвертированные повторы[англ.], представляющие палиндромные последовательности нуклеотидов на обоих концах генома[6].
Репликация и транскрипция
[править | править код]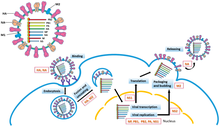
РНК-зависимая РНК-полимераза (RdRp) использует негативную цепь генома в качестве матрицы для синтеза комплиментарной положительной цепи. Репликация оц(-)РНК-вирусов осуществляется с помощью RdRp, которая инициирует репликацию путем связывания с лидерной последовательностью на 3'-конце генома. После этого, RdRp игнорирует все сигналы терминации транскрипции на негативной цепи и синтезирует полную копию генома.[7]. Репликация начинается, пока РНК вируса находится внутри нуклеокапсида, а RdRp перемещается по геному раскрывая капсид во время репликации. По мере того, как новые нуклеотидные последовательности синтезируются RdRp, белки капсида собираются на новых молекулах[8].
Транскрипция мРНК с геномной РНК происходит по той же схеме, что и создание комплиментарной положительной цепи геномной РНК. На лидерной последовательности, RdRp синтезирует 5'-концевую (обычно произносится «пять штрих конец») лидерную РНК, оканчивающуюся тремя фосфатными группами, затем, в случае подтипа Haploviricotina, кэпирует её 5'-конец или, в случает подтипа Polyploviricotina, использует отрыв кэпа[англ.] от мРНК клетки-хозяина и соединяет его с вирусными мРНК, после чего эти мРНК могут быть транслированы на рибосомах клеток хозяина.[9][10][11]
После кэпирования мРНК RdRp инициирует транскрипцию в старт-кодоне, а затем прекращает транскрипцию по достижении стоп-кодона. В конце транскрипции RdRp синтезирует полиадинелированный хвост (полиА-хвост), состоящий из сотен остатков аденина на 3'-конце мРНК, что может происходить путем "пробуксовывания" (синтеза цепи нуклеотидов без продвижения по матрице) RdRp на последовательности урацилов. После того, как полиА-хвост синтезирован, мРНК высвобождается из комплекса с RdRp. В геномах, которые кодируют более одного транскрипта, RdRp может продолжить сканирование цепи родительской РНК до следующего стартового кодона, чтобы продолжить транскрипцию[9][12].
Некоторые оц(-)РНК-вирусы являются двухполярными[англ.], что означает, что и отрицательная (геномная) цепь РНК, и положительная (антигеномная) цепь по отдельности кодируют разные белки. При транскрипции двухполярных вирусов выполняется два раунда: во-первых, мРНК считываются с непосредственно с геномной РНК; во-вторых, мРНК считываются с оц(+)РНК антигенома. Все двухполярные вирусы содержат структуру типа шпильки для остановки транскрипции после того, как будут транскрибированы мРНК, кодирующие белки[13].
Строение
[править | править код]
Эволюция
[править | править код]Сегментация генома является характерной чертой для многих оц(-)РНК-вирусов, число сегментов варьируется от одного, что является типичным для представителей отряда Mononegavirales, до десяти сегментов, как в случае генома Tilapia lake virus[6][14]. Нет четкой тенденции к увеличению или уменьшению сегментирования генома оц(-)РНК-вирусов с течением времени. По-видимому число сегментов является гибким признаком, поскольку он эволюционировал независимо во многих случаях. Большинство представителей подтипа Haploviricotina имеет несегментированный геном, в товремя как все члены подтипа Polyploviricotina с сегментированным геномом[6][8].
Поскольку оц(-)RNA-вирусам необходимо транскрибировать свои геномы в мРНК, перед их трансляцией, некоторый контроль над экспрессией генов может происходить на этом этапе транскрипции. С одной (-)цепи РНК может быть транскрибировано несколько мРНК, при этом первая мРНК (транскрипция которой начинается ближе всех к 3’-концу) будет представлена в большей концентрации, а последняя (5’-концевая) мРНК в наименьшей концентрации. То есть в зависимости от расположения старта транскрипции мРНК в геноме вируса создается транскрипционный градиент. Следовательно, возможно, что способность лучше контролировать экспрессию генов посредством контроля транскрипции сама по себе представляет собой причину, по которой в первую очередь возникали геномы с антисмысловой (-)РНК. В этом отношении важно, что геномы несегментированных оц(-)РНК– вирусов обладают высококонсервативным порядком генов, составляют одну группу при построении филогенетических деревьев на основе последовательностей полимераз, и могут быть легко классифицированы в пределах группы Mononegavirales. Более того, при этом способе организации генома, порядок генов, по-видимому, зависит от требуемого количества белкового продукта этих генов, так что первые гены расположенные ближе к 3’-концу кодируют белки нуклеокапсида, а гены расположенные на 5’-конце, кодируют РНК-полимеразу. Это подтверждает предположение, что это адаптация для облегчения контроля экспрессии генов[15].
Филогенетический анализ
[править | править код]
Филогенетический анализ основанный на последовательности RdRp, показывает, что оц(-)РНК-вирусы произошли от общего предка, и что они, вероятно, являются сестринской кладой по отношению к реовирусам, которые являются вирусами с двухцепочечной РНК. Внутри этого типа есть две чёткие ветви, относящиеся к двум подтипам, в зависимости от того, синтезирует ли RdRp кэп на вирусной мРНК или осуществляет отрыв кэпа[англ.] от мРНК клетки-хозяина и присоединяет его к мРНК вируса[3][4].
Вирусы, которые инфицируют членистоногих, по видимому являются базальной группой и являются предками всех других оц(-)РНК-вирусов внутри этого типа. Членистоногие часто обитают вместе большими группами, что позволяет вирусам легко передаваться между ними. Со временем это привело к большому уровню разнообразия оц(-)РНК-вирусов насекомых. Хотя членистоногие и являются хозяевами большого количества вирусов, существуют разногласия относительно степени передачи вирусов между видами членистоногих[5][6].
Оц(-)РНК-вирусы растений и позвоночных обычно генетически родственны с вирусами инфицирующими членистоногих. Более того, большинство таких вирусов обнаруживается у видов растений и животных, которые взаимодействуют с членистоногими. Таким образом, членистоногие служат как основными хозяевами, так и переносчиками вирусов. С этой точки зрения вирусы могут быть разделены на тех, которые используют членистоногих в качестве переносчика и тех, которые имеют свое происхождение от вирусов членистоногих, но теперь воспроизводятся в клетках позвоночных, передаваясь без их помощи[6].
Классификация
[править | править код]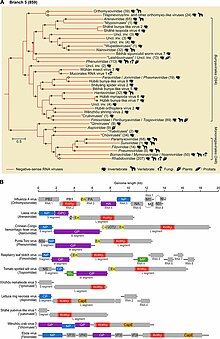
На июль 2021 года в Negarnaviricota включают 2 подтипа и 6 классов, 3 из которых являются монотипными вплоть до родов, а 2 — до порядков[2]:
- Подтип Haploviricotina, который включает оц(-)РНК-вирусы, кодирующие RdRp, которая синтезирует структуру кэпа на вирусных мРНК, и которые обычно имеют несегментированный геном[источник не указан 1196 дней]
- Класс Chunquiviricetes
- Порядок Muvirales
- Семейство Qinviridae
- Род Yingvirus
- Семейство Qinviridae
- Порядок Muvirales
- Класс Milneviricetes
- Порядок Serpentovirales
- Семейство Aspviridae
- Род Ophiovirus
- Семейство Aspviridae
- Порядок Serpentovirales
- Класс Monjiviricetes
- Порядок Jingchuvirales
- Порядок Mononegavirales
- Класс Yunchangviricetes
- Порядок Goujianvirales
- Семейство Yueviridae
- Род Yuyuevirus
- Семейство Yueviridae
- Порядок Goujianvirales
- Класс Chunquiviricetes
- Подтип Polyploviricotina, который содержит оц(-)РНК-вирусы, кодирующие RdRp, которая отрезает кэп от мРНК клеток-хозяев и использует их, как кэп вирусных мРНК. Эти вирусы имеют сегментированный геном[источник не указан 1196 дней]
- Класс Ellioviricetes
- Порядок Bunyavirales
- Класс Insthoviricetes
- Порядок Articulavirales
- Класс Ellioviricetes
См. также
[править | править код]Примечания
[править | править код]- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ 1 2 Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV). (Дата обращения: 31 июля 2021).
- ↑ 1 2 3 4 Wolf Y., Krupovic M., Zhang Y. Z., Maes P., Dolji V., Koonin E. V. Megataxonomy of negative-sense RNA viruses (англ.) (docx). ICTV (21 августа 2017). Дата обращения: 6 августа 2020. Архивировано 16 ноября 2021 года.
- ↑ 1 2 Wolf YI, Kazlauskas D, Iranzo J, Lucia-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Kooning EV (2018-11-27). "Origins and Evolution of the Global RNA Virome". mBio. 9 (6): e02329-18. doi:10.1128/mBio.02329-18. PMC 6282212. PMID 30482837.
- ↑ 1 2 Käfer S, Paraskevopoulou S, Zirkel F, Wieseke N, Donath A, Petersen M, Jones TC, Liu S, Zhou X, Middendorf M, Junglen S, Misof B, Drosten C (2019-12-12). "Re-assessing the diversity of negative strand RNA viruses in insects". PLOS Pathog. 15 (12): e1008224. doi:10.1371/journal.ppat.1008224. PMC 6932829. PMID 31830128.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ 1 2 3 4 5 Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (2015-01-29). "Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses". eLife. 4 (4): e05378. doi:10.7554/eLife.05378. PMC 4384744. PMID 25633976. Архивировано 26 мая 2021. Дата обращения: 6 августа 2020.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Negative stranded RNA virus replication. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020. Архивировано 5 мая 2021 года.
- ↑ 1 2 Luo M, Terrel JR, Mcmanus SA (2020-07-30). "Nucleocapsid Structure of Negative Strand RNA Virus". Viruses. 12 (8): 835. doi:10.3390/v12080835. PMC 7472042. PMID 32751700.
- ↑ 1 2 Negative-stranded RNA virus transcription. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020. Архивировано 23 мая 2021 года.
- ↑ Cap snatching. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020. Архивировано 8 октября 2020 года.
- ↑ Kuhn JH, Wolf YI, Krupovic M, Zhang YZ, Maes P, Dolja VV, Koonin EV (February 2019). "Classify viruses - the gain is worth the pain". Nature. 566 (7744): 318—320. Bibcode:2019Natur.566..318K. doi:10.1038/d41586-019-00599-8. PMID 30787460. Архивировано 7 ноября 2020. Дата обращения: 6 августа 2020.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Negative-stranded RNA virus polymerase stuttering. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020. Архивировано 10 мая 2021 года.
- ↑ Ambisense transcription in negative stranded RNA viruses. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020. Архивировано 26 февраля 2021 года.
- ↑ Bacharach E, Mishra N, Briese T, Zody MC, Kembou Tsofack JE, Zamostiano R, Berkowitz A, Ng J, Nitido A, Corvelo A, Toussaint NC, Abel Nielsen SC, Hornig M, Del Pozo J, Bloom T, Ferguson H, Eldar A, Lipkin WI (2016-04-05). "Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia". mBio. 7 (2): e00431-16. doi:10.1128/mBio.00431-16. PMC 4959514. PMID 27048802.
- ↑ Holmes, 2009.
Литература
[править | править код]- Ward, C. W. (1993). "Progress towards a higher taxonomy of viruses". Research in Virology. 144 (6): 419—453. doi:10.1016/S0923-2516(06)80059-2. PMC 7135741. PMID 8140287.
- Holmes E.C. The Evolution and Emergence of RNA Viruses. (англ.). — Oxford: Oxford University Press, 2009.