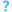فيروسات الأورام الحليمية

هذه مقالة غير مراجعة. (مايو 2020) |
فيروس الأورام الحليمية | |
|---|---|
صورة بالمجهر الإلكتروني لفيروس الأورام الحليمية | |
| المرتبة التصنيفية | فصيلة[1][2] |
| تصنيف الفيروسات | |
| المجموعة: | (dsDNA) I مجموعة |
| الفصيلة: | فيروسات الأورام الحليمية |
| الاسم العلمي | |
| Papillomaviridae[1][2] | |
| Genera | |
| فيروس تورامي ألفا فيروس تورامي بيتا | |
| تعديل مصدري - تعديل | |
فيروسات الأورام الحليمية (الاسم العلمي: Papillomaviridae) عائلةٌ من فيروسات الدنا غير المغلفة.[8] عُرفت مئات الأنواع من هذه الفيروسات التي يُشار إليها عادةً بكلمة «أنماط»،[9] ووُجد أنها تصيب جميع الثدييات المدروسة بدقة،[9] إضافةً إلى الفقاريات الأخرى مثل الطيور والأفاعي والسلاحف والأسماك.[10][11][12] تعتمد الإصابة بأغلب أنواع الفيروسات الحليمية على النمط، وتكون إما غير عرضية (أغلب فيرسات بيتا) أو تسبب أورامًا حميدةً صغيرة الحجم تُعرف بالأورام الحليمية أو الثآليل (مثل فيروسات الورم الحليمي البشري 1 و6 و11). تحمل بعض الأورام الحليمية الناجمة عن العدوى ببعض الأنماط مثل الفيروسات الحليمية 16 و18 خطر التحول إلى الخباثة.[13]
تميل فيروسات الورم الحليمي إلى الانتحاء نحو المضيف والنسج، ويُعتقد أنها نادرًا ما تنتقل بين الأنواع.[14] تنتسخ هذه الفيروسات حصريًا في الطبقة القاعدية من النسج السطحية في الجسم. تصيب جميع أنماط الفيروسات الحليمية المعروفة نسيجًا سطحيًا محددًا في الجسم، وعادةً ما يكون الجلد أو الظهارة المخاطية التناسلية أو ظهارة الشرج والفم والطرق الهوائية.[15] يميل الفيروس الحليمي نمط 1 لإصابة أخمصي القدمين، بينما يصيب النمط 2 راحتي اليدين حيث قد يسببان الثآليل. هناك من وصف وجود فيروسات الدنا الحليمية في الدم وخلايا الدم المحيطي وحيدة النواة.
عُرفت فيروسات الأورام الحليمية للمرة الأولى في أوائل القرن العشرين، حين ثبت أن الثآليل والأورام الحليمية قابلة للانتقال بين الأفراد عبر عامل معدٍ قابل للارتشاح. في عام 1935، مضى فرانسيس بيتون روس -الذي سبق وكشف وجود فيروسات الأورام العفلية المسببة للسرطان في الدجاج- إلى إثبات أن الفيروسات الحليمية قد تسبب سرطان الجلد في الأرانب المصابة. كان هذا أول برهان على أن الفيروسات قادرة على إحداث السرطان في الثدييات.
تصنيف فيروسات الأورام الحليمية
[عدل]كشف الباحثون عن ما يزيد على 100 نوع من الفيروسات الحليمية، لكن اللجنة الدولية لتصنيف الفيروسات تعترف بعدد أقل،[16] إذ صنفتها إلى 53 جنسًا عام 2019.[17][18][19] تملك جميع الفيروسات الحليمية تنظيمات جينوميةً متشابهةً، ويحتوي أي زوج منها على خمسة جينات متماثلة على الأقل، لكن تسلسل النكليوتيدات قد يختلف بما يزيد عن 50%. مكنت خوارزميات تطور السلالات التي تسمح بمقارنة التنادد بينها من تشكيل شجرة تطور السلالات التي أبدت طوبولوجيا متشابهة مستقلة عن التحليل الجيني.[20]
تقترح دراسات تطور السلالات بقوة أن فيروسات الورم الحليمي تتطور بشكل طبيعي مع أنواعها المضيفة من الطيور والثدييات، لكن التشعب التكيفي والأحداث حيوانية المنشأ والتأشيب المورثي قد تؤثر أيضًا على تنوعها. يبدو أن التنظيمات الجينومية الأساسية صانت نفسها لفترة تتجاوز 100 مليون عام، وهذه المقارنات بين التسلسلات الوراثية أسست لتصنيف فيروسات الورم الحليمي التي تعترف بها اللجنة الدولية لتصنيف الفيروسات اليوم رسميًا.
تشكل مجموعة الفيروسات الحليمية عائلة فيروسات الورم الحليمي التي انفصلت عن عائلة الفيروسات التورامية وألغت بذلك مصطلح فيروسات البابوفا الذي كان يشمل كلا العائلتين. يعرف الجنس بأنه الفرع الرئيسي من شجرة تطور السلالات، ويُشار إليه بالأحرف اليونانية. تسمى الفروع الصغرى «بالأنواع»، وهي تجمع أنماط الفيروسات الحليمية المختلفة جينيًا دون بيان الاختلافات الحيوية المعروفة.[21]
لا يؤثر نظام التصنيف الجديد على التعريف والتوصيف التقليدي لأنماط الفيروسات الحليمية وأنواعها المعزولة المستقلة مع اختلافاتها الجينية الصغرى، والتي يُشار إليها «بالأنماط الفرعية» أو «المتغايرات»، وجميعها تُصنف تحت مستوى «أنواع». من المقترح إضافة مجموعات تطور السلالات إلى مستوًى تصنيفي أعلى.[22]
قد يحتاج هذا التصنيف إلى تنقيح في ضوء وجود فيروسات هجينة بين الحليمية والتورامية.[23] وُصفت المزيد من أنواع هذه الفيروسات، وأمكن عزل فيروس الورم الحليمي الخاص بسمك الدنيس من الأسماك المضيفة.[24]
فيروسات الأورام الحليمية البشرية
[عدل]تمكن العلماء من كشف التسلسل الوراثي الكامل لأكثر من 170 نوعًا من فيروسات الورم الحليمي البشري.[25] قُسمت هذه الفيروسات إلى خمسة أجناس: ألفا وبيتا وغاما ومو ونو. هناك 200 فيروس إضافي على الأقل بانتظار التسلسل الوراثي والتصنيف.
فيروسات الأروام الحليمية الحيوانية
[عدل]تميل فيروسات الأورام الحليمية المفردة إلى التأقلم إلى حد كبير مع الانتساخ في نوع حيواني مفرد.[26] في إحدى الدراسات، أخذ الباحثون مسحةً من جلد الجبين لأنواع مختلفة من حيوانات الحدائق، واستخدموا تفاعل البوليميراز المتسلسل لتضخيم أي كمية من الدنا الخاص بالفيروسات الحليمية التي قد تكون قابعة في هذا النسيج. وجد المؤلفون دليلًا ضعيفًا على الانتقال بين الأنواع رغم كشف العديد من التسلسلات الوراثية الخاصة بهذه الفيروسات في الدراسة. كُشفت إصابة عابرة لدى أحد الحراس بفيروس الورم الحليمي الخاص بالشمبانزي، ولكن ذلك قد يكون نتيجة العدوى السطحية لجلد الحارس وليس بسبب العدوى الفعالة.
قد يسبب فيروس الورم الحليمي الخاص بالأرنب قطني الذيل (سي بّي آر في) ثآليل ناتئة لدى المضيف الأصلي، وهو جنس الأرنب الأمريكي الشمالي (سلفيلاغوس Sylvilagus). قد تكون هذه الثآليل القرنية الأساس الأصلي للأسطورة المدنية حول الأرنب الأمريكي ذو قرن الوعل «الجاكالوب» (من الفلكلور الأمريكي الشمالي) و«الوولبردنغر» الأوروبي (من الفلكلور الألماني).
قد تعاني الأرانب الأوروبية المحلية (جنس أوريكتولاغوس Oryctolagus) من إصابة عابرة بفيروس الأرنب قطني الذيل في ظروف المخبر، لكن هذه الأرانب لا تنتج سلالة فيروسية معدية، وبالتالي تُعد مضيفًا عرضيًا أو انتهائيًا لسلسلة عدوى هذا الفيروس.[27][28]
وُثق الانتقال بين الأنواع بفيروس الورم الحليمي البقري نمط 1.[29] يحرض هذا الفيروس ظهور ثآليل ليفية جلدية كبيرة في مضيفه الطبيعي (الأبقار). قد تؤدي عدوى الخيول، التي تمثل مضيفًا عرضيًا للفيروس، إلى تطور أورام حميدة تُعرف بالورم السرطاوي (داء الساركويد). تنبع الأهمية الزراعية لفيروس الورم الحليمي البقري من إنجاحه للجهد المبذول في تطوير لقاح مضاد للفيروس.
كشفت بعض التقارير عن وجود فيروسات حليمية في القوارض الأصغر، مثل القداد السوري والجرذ الأفريقي عديد الأثداء وفأر الحصاد الأوروآسيوي.[30] لم يُكشف بعد أي فيروس قادر على عدوى فئران التجارب، لكن لا يمكن اعتبار هذه المعلومة دقيقةً. شكل نقص نماذج الفئران المطاوعة عقبةً كبيرةً أمام الاستقصاءات المخبرية على فيروسات الورم الحليمي.
هناك أربعة فيروسات حليمية عُرفت بقدرتها على إصابة الطيور: فيروس الحسون الظالم الحليمي نمط 1، وفيروس الدراج أصفر العنق الحليمي نمط 1، وفيروس الببغاء الأفريقي الحليمي نمط 1، وفيروس البطريق الآديلي الحليمي نمط 1.[31] تحمل جميع هذه الفيروسات المورثة إي 9 غير معروفة الوظيفة، ما يقترح أصلها المشترك.
التطور
[عدل]يُعتقد أن تطور فيروسات الورم الحليمي بطيء مقارنةً بالأنماط الفيروسية الأخرى، لكن لا تتوافر حاليًا إجراءات تجريبية لإثبات هذه الفرضية. قد يعود هذا إلى جينوم هذه الفيروسات المؤلف من شريط دنا مزدوج مستقر وراثيًا ينتسخ بدقة اعتمادًا على آليات الخلية المضيفة في انتساخ الدنا.
يعتقد الباحثون أن هذه الفيروسات تخضع للتطور المشترك مع أنواع محددة من الحيوانات المضيفة على مدى سنوات عديدة رغم وجود دليل قوي يدحض هذه الفرضية.[20][32] أحد الأمثلة المقتضبة على ذلك تطور فيروس الورم الحليمي نمط 16 بشكل بسيط بعد انتشار البشر في بقاع الكوكب، والآن يختلف بين المناطق الجغرافية المتنوعة بأسلوب يعكس تاريخ الهجرة البشرية.[33][34]
تنتقل أنماط الفيروسات الحليمية البشرية الجلدية أحيانًا بين أفراد العائلة خلال فترة الحياة، لكن يجب الانتباه أيضًا إلى المانحين الآخرين في عملية الانتقال الفيروسي.[35]
تختلف أنماط فيروس الورم الحليمي البشري الأخرى مثل النمط 13 نسبيًا بين البشر. يشابه تسلسل الفيروس 13 تسلسل فيروس البونوبو (المعروف أيضًا بشمبانزي بيغمي أو الشمبانزي القزم).[36] من غير الواضح فيما إذا كانت هذه التشابهات نتيجة انتقال حديث بين الأنواع أو بسبب تغير النمط 13 بشكل بسيط خلال ستة ملايين سنة سابقة أو ما يقاربها منذ افتراق الجنس البشري عن البونوبو.[34]
يُقدر أن أقرب سلف مشترك لهذه المجموعة من الفيروسات وُجد منذ نحو 424 مليون سنة.[37] هناك خمسة أجناس رئيسة تصيب البشر (ألفا، بيتا، غاما، مو، نو). تطور السلف المشترك الأقرب لهذه الأجناس منذ نحو 49.7 إلى 58,5 مليون سنة. يقدر أن أقرب سلف لجنس غاما تطور بين 45.3 إلى 67.5 مليون سنة.[38]
اقرأ أيضًا
[عدل]المراجع
[عدل]- ^ ا ب ج International Committee on Taxonomy of Viruses, ed. (30 Jun 2014), ICTV Master Species List 2013 v2 (بالإنجليزية), QID:Q18810383
- ^ ا ب ج International Committee on Taxonomy of Viruses, ed. (12 Jun 2015), ICTV Master Species List 2014 v4 (بالإنجليزية), QID:Q24716610
- ^ International Committee on Taxonomy of Viruses, ed. (26 May 2016), ICTV Master Species List 2015 v1 (بالإنجليزية), QID:Q24571893
- ^ International Committee on Taxonomy of Viruses, ed. (25 May 2017), ICTV Master Species List 2016 v1.3 (بالإنجليزية), QID:Q29000566
- ^ International Committee on Taxonomy of Viruses, ed. (17 Oct 2018), ICTV Master Species List 2018a v1 (بالإنجليزية), QID:Q57700017
- ^ International Committee on Taxonomy of Viruses, ed. (8 Mar 2019), ICTV Master Species List 2018b.v2 (بالإنجليزية), QID:Q62075759
- ^ International Committee on Taxonomy of Viruses, ed. (13 Mar 2018), ICTV Master Species List 2017 v1 (بالإنجليزية), QID:Q51526638
- ^ Van Doorslaer، K؛ Chen، Z؛ Bernard، HU؛ Chan، PKS؛ DeSalle، R؛ Dillner، J؛ Forslund، O؛ Haga، T؛ McBride، AA؛ Villa، LL؛ Burk، RD؛ Ictv Report، Consortium (أغسطس 2018). "ICTV Virus Taxonomy Profile: Papillomaviridae". The Journal of General Virology. ج. 99 ع. 8: 989–990. DOI:10.1099/jgv.0.001105. PMC:6171710. PMID:29927370.
- ^ ا ب de Villiers EM، Fauquet C، Broker TR، Bernard HU، zur Hausen H (يونيو 2004). "Classification of papillomaviruses". Virology. ج. 324 ع. 1: 17–27. DOI:10.1016/j.virol.2004.03.033. PMID:15183049.
- ^ Herbst LH، Lenz J، Van Doorslaer K، Chen Z، Stacy BA، Wellehan JF، Manire CA، Burk RD (يناير 2009). "Genomic characterization of two novel reptilian papillomaviruses, Chelonia mydas papillomavirus 1 and Caretta caretta papillomavirus 1". Virology. ج. 383 ع. 1: 131–5. DOI:10.1016/j.virol.2008.09.022. PMID:18973915.
- ^ Drury SE، Gough RE، McArthur S، Jessop M (ديسمبر 1998). "Detection of herpesvirus-like and papillomavirus-like particles associated with diseases of tortoises". The Veterinary Record. ج. 143 ع. 23: 639. PMID:9881444.
- ^ Lange CE، Favrot C، Ackermann M، Gull J، Vetsch E، Tobler K (سبتمبر 2011). "Novel snake papillomavirus does not cluster with other non-mammalian papillomaviruses". Virology Journal. ج. 8: 436. DOI:10.1186/1743-422X-8-436. PMC:3179961. PMID:21910860.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Muñoz N، Castellsagué X، de González AB، Gissmann L (أغسطس 2006). "Chapter 1: HPV in the etiology of human cancer". Vaccine. 24 Suppl 3 ع. 3: S3/1–10. DOI:10.1016/j.vaccine.2006.05.115. PMID:16949995.
- ^ Mistry N، Wibom C، Evander M (أكتوبر 2008). "Cutaneous and mucosal human papillomaviruses differ in net surface charge, potential impact on tropism". Virology Journal. ج. 5: 118. DOI:10.1186/1743-422X-5-118. PMC:2571092. PMID:18854037.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Doorbar J (مارس 2005). "The papillomavirus life cycle". Journal of Clinical Virology. 32 Suppl 1: S7–15. DOI:10.1016/j.jcv.2004.12.006. PMID:15753007.
- ^ Kocjan BJ، Hosnjak L، Seme K، Poljak M (مايو 2013). "Complete Genome Sequence of a Novel Human Betapapillomavirus, HPV-159". Genome Announcements. ج. 1 ع. 3: e00298–13. DOI:10.1128/genomeA.00298-13. PMC:3668007. PMID:23723399.
- ^ "Virus Taxonomy: 2018b Release". International Committee on Taxonomy of Viruses. فبراير 2019. مؤرشف من الأصل في 2020-11-21. اطلع عليه بتاريخ 2019-03-26.
- ^ "Virus Taxonomy: 2014 Release". ICTV. مؤرشف من الأصل في 2017-02-10. اطلع عليه بتاريخ 2015-06-15.
- ^ Bernard HU، Burk RD، Chen Z، van Doorslaer K، zur Hausen H، de Villiers EM (مايو 2010). "Classification of papillomaviruses (PVs) based on 189 PV types and proposal of taxonomic amendments". Virology. ج. 401 ع. 1: 70–9. DOI:10.1016/j.virol.2010.02.002. PMC:3400342. PMID:20206957.
- ^ ا ب Gottschling M، Stamatakis A، Nindl I، Stockfleth E، Alonso Á، Bravo IG (2007). "Multiple evolutionary mechanisms drive papillomavirus diversification". Molecular Biology and Evolution. ج. 24 ع. 5: 1242–58. DOI:10.1093/molbev/msm039. PMID:17344207.
- ^ Campo MS، المحرر (2006). Papillomavirus Research: From Natural History To Vaccines and Beyond. Caister Academic Press. ISBN:978-1-904455-04-2. [1]. مؤرشف من الأصل في 2016-05-22.
- ^ Bravo IG، de Sanjosé Llongueras S، Gottschling M (2010). "The clinical importance of knowledge about papillomavirus evolution". Trends in Microbiology. ج. 18 ع. 10: 432–8. DOI:10.1016/j.tim.2010.07.008. PMID:20739182.
- ^ Rector A، Van Ranst M (أكتوبر 2013). "Animal papillomaviruses". Virology. ج. 445 ع. 1–2: 213–23. DOI:10.1016/j.virol.2013.05.007. PMID:23711385.
- ^ López-Bueno A، Mavian C، Labella AM، Castro D، Borrego JJ، Alcami A، Alejo A (أكتوبر 2016). "Concurrence of Iridovirus, Polyomavirus, and a Unique Member of a New Group of Fish Papillomaviruses in Lymphocystis Disease-Affected Gilthead Sea Bream". Journal of Virology. ج. 90 ع. 19: 8768–79. DOI:10.1128/JVI.01369-16. PMC:5021401. PMID:27440877.
- ^ Chouhy D، Bolatti EM، Pérez GR، Giri AA (نوفمبر 2013). "Analysis of the genetic diversity and phylogenetic relationships of putative human papillomavirus types". The Journal of General Virology. ج. 94 ع. Pt 11: 2480–8. DOI:10.1099/vir.0.055137-0. hdl:2133/9862. PMID:23997181.
- ^ Antonsson A، Hansson BG (ديسمبر 2002). "Healthy skin of many animal species harbors papillomaviruses which are closely related to their human counterparts". Journal of Virology. ج. 76 ع. 24: 12537–42. DOI:10.1128/JVI.76.24.12537-12542.2002. PMC:136724. PMID:12438579.
- ^ Holliday، Chuck. "Prof. Chuck Holliday's www page at Lafayette College » Jackalopes". مؤرشف من الأصل في 2014-07-18. اطلع عليه بتاريخ 2014-07-13.
- ^ Christensen ND (2005). "Cottontail rabbit papillomavirus (CRPV) model system to test antiviral and immunotherapeutic strategies". Antiviral Chemistry & Chemotherapy. ج. 16 ع. 6: 355–62. DOI:10.1177/095632020501600602. PMID:16331841.
- ^ Coggins LW، Ma JQ، Slater AA، Campo MS (يونيو 1985). "Sequence homologies between bovine papillomavirus genomes mapped by a novel low-stringency heteroduplex method". Virology. ج. 143 ع. 2: 603–11. DOI:10.1016/0042-6822(85)90398-8. PMID:2998027.
- ^ Van Ranst M، Tachezy R، Pruss J، Burk RD (يونيو 1992). "Primary structure of the E6 protein of Micromys minutus papillomavirus and Mastomys natalensis papillomavirus". Nucleic Acids Research. ج. 20 ع. 11: 2889. DOI:10.1093/nar/20.11.2889. PMC:336941. PMID:1319576.
- ^ Varsani A، Kraberger S، Jennings S، Porzig EL، Julian L، Massaro M، Pollard A، Ballard G، Ainley DG (يونيو 2014). "A novel papillomavirus in Adélie penguin (Pygoscelis adeliae) faeces sampled at the Cape Crozier colony, Antarctica". The Journal of General Virology. ج. 95 ع. Pt 6: 1352–65. DOI:10.1099/vir.0.064436-0. PMID:24686913. S2CID:206218507.
- ^ Gottschling M، Göker M، Stamatakis A، Bininda-Emonds OR، Nindl I، Bravo IG (يوليو 2011). "Quantifying the phylodynamic forces driving papillomavirus evolution". Molecular Biology and Evolution. ج. 28 ع. 7: 2101–13. DOI:10.1093/molbev/msr030. PMID:21285031.
- ^ Ho L، Chan SY، Burk RD، Das BC، Fujinaga K، Icenogle JP، Kahn T، Kiviat N، Lancaster W، Mavromara-Nazos P (نوفمبر 1993). "The genetic drift of human papillomavirus type 16 is a means of reconstructing prehistoric viral spread and the movement of ancient human populations". Journal of Virology. ج. 67 ع. 11: 6413–23. DOI:10.1128/JVI.67.11.6413-6423.1993. PMC:238076. PMID:8411343.
- ^ ا ب Calleja-Macias IE، Villa LL، Prado JC، Kalantari M، Allan B، Williamson AL، Chung LP، Collins RJ، Zuna RE، Dunn ST، Chu TY، Cubie HA، Cuschieri K، von Knebel-Doeberitz M، Martins CR، Sanchez GI، Bosch FX، Munoz N، Bernard HU (نوفمبر 2005). "Worldwide genomic diversity of the high-risk human papillomavirus types 31, 35, 52, and 58, four close relatives of human papillomavirus type 16". Journal of Virology. ج. 79 ع. 21: 13630–40. DOI:10.1128/JVI.79.21.13630-13640.2005. PMC:1262609. PMID:16227283.
- ^ Gottschling M، Göker M، Köhler A، Lehmann MD، Stockfleth E، Nindl I (2009). "Cutaneotropic β-/γ-HPV types are rarely shared between family members". Journal of Investigative Dermatology. ج. 129 ع. 10: 2427–34. DOI:10.1038/jid.2009.94. PMID:19516265.
- ^ Van Ranst M، Fuse A، Fiten P، Beuken E، Pfister H، Burk RD، Opdenakker G (أكتوبر 1992). "Human papillomavirus type 13 and pygmy chimpanzee papillomavirus type 1: comparison of the genome organizations". Virology. ج. 190 ع. 2: 587–96. DOI:10.1016/0042-6822(92)90896-W. PMID:1325697.
- ^ Willemsen A, Bravo IG (2019) Origin and evolution of papillomavirus (onco)genes and genomes. Philos Trans R Soc Lond B Biol Sci. 374(1773):20180303
- ^ Murahwa AT, Nindo F, Onywera H, Meiring TL, Martin DP, Williamson AL (2019) Evolutionary dynamics of ten novel Gamma-PVs: insights from phylogenetic incongruence, recombination and phylodynamic analyses. BMC Genomics 20(1):368