Empalme alternativo

El empalme alternativo (alternative splicing en inglés), ajuste alternativo o splicing alternativo, permite obtener a partir de un transcrito primario de ARNm o pre-ARNm distintas isoformas de ARNm y proteínas, las cuales pueden tener funciones diferentes y a menudo opuestas. Este proceso ocurre principalmente en eucariotas, aunque también puede observarse en virus.[1]
Introducción
[editar]Muchos genes están empalmados alternativamente en formas específicas de tejidos, reguladas en el desarrollo y en respuesta a hormonas, proporcionando un mecanismo adicional para la regulación de la expresión génica. Al transcribirse el ADN a ARNm se obtiene un transcrito primario de ARN o pre-ARNm que abarca intrones y exones.[2] Para que este pre-ARNm dé lugar a un ARNm debe sufrir un proceso de maduración del ARNm, que consiste, básicamente, en eliminar todos los intrones. Sin embargo, los intrones y exones no siempre están determinados durante el proceso de ajuste. La selección de los sitios de ajuste se lleva a cabo por residuos de serina/arginina de ciertas proteínas conocidas como proteínas SR.
Un hallazgo crítico con respecto a la prevalencia de splicing alternativo fue que la mayoría de los genes humanos producen una amplia variedad de ARNm que a su vez codifican proteínas distintas.[3] Los científicos estiman que el 15-60 % de las enfermedades genéticas humanas implican mutaciones de empalme, ya sea a través de la mutación directa de las señales del sitio de empalme o a través de la interrupción de otros componentes de la vía de empalme. Es por esta razón que tanto las zonas aceptoras como donadoras de splicing son consideradas regiones altamente intolerantes a la variación dada su esencialidad.[4] Por lo tanto, la comprensión de qué información en pre-mRNAs determina splicing alternativo y cómo las células regulan splicing alternativo es de importancia crítica.[5]
Tipos de empalme alternativo
[editar]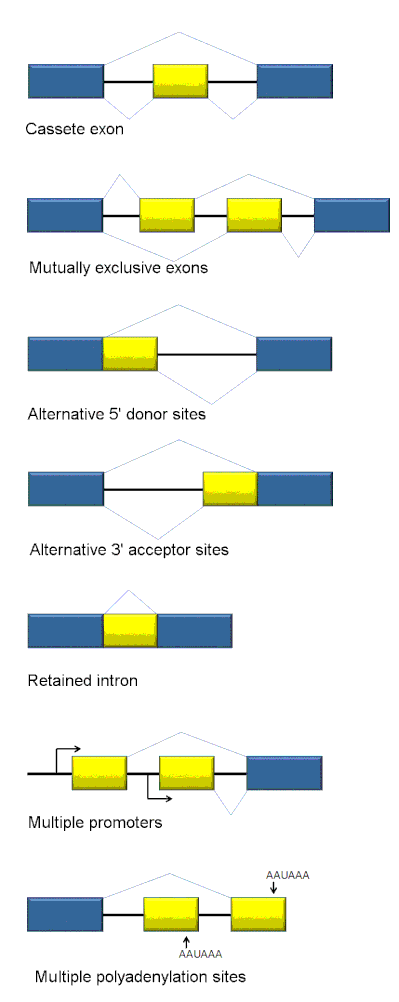
Los tipos de empalme alternativo incluyen el uso de sitios alternativos de empalme en 5' y 3', exones de casete, intrones retenidos y exones mutuamente exclusivos (Figura 1).[6]
- Cassete del exón: en este caso, un exón puede ser empalmado fuera de la transcripción primaria o retenido. Este es el modo más común en pre-mRNAs de mamíferos.
- Exones mutuamente exclusivos: Uno de los dos exones se conserva en los ARNm después del empalme, pero no ambos.
- Sitio alternativo 5': Se usa una unión de empalme alternativa para el sitio 5' (sitio donante), cambiando el límite 3' del exón aguas arriba.
- Sitio alternativo 3': Se usa una unión de empalme alternativa para el sitio 3' (sitio aceptor), cambiando el límite 5' del exón aguas abajo.
- Retención de Intrón: Una secuencia puede ser empalmada como un exón o simplemente retenida. Esto se distingue de la omisión de exón porque la secuencia retenida no está flanqueada por intrones. Si el intrón retenido está en la región de codificación, el intrón debe codificar los aminoácidos en el marco con los exones vecinos, o un codón de terminación o un cambio en el marco de lectura hará que la proteína sea no funcional. Este es el modo más raro en los mamíferos.
- Selección de promotores alternativos: este es el único método que da lugar a un dominio N-terminal alternativo. En este caso, cada promotor puede dar lugar a un juego de exones diferentes.
- Selección de sitios de poliadenilación alternativos: este es el único método que da lugar a un dominio C-terminal alternativo. En este caso, cada sitio de poliadenilación puede dar lugar a un juego de exones diferentes.[6]
Importancia en genética molecular
[editar]El splicing alternativo invalida la vieja teoría «un gen, una proteína», siendo por tanto necesaria la información externa para decidir que polipéptido será sintetizado. Al mismo tiempo este sistema permite almacenar la información de forma más económica. Así, por ejemplo, este sistema permite obtener varias proteínas a partir de una única secuencia de ADN.[3]
Algunos investigadores han sugerido que este sistema permitiría obtener nuevas proteínas cambiando los mecanismos de regulación. También se ha sugerido que este sistema permitiría una evolución más rápida.
Existe una creencia que afirma que los sitios alternativos de splicing son responsables de la complejidad de los humanos; afirmando que los genes humanos tienen más sitios alternativos de splicing. Sin embargo un estudio de David Brett y colaboradores[2] afirma que no existen diferencias significativas entre el número de sitios de splicing alternativos de humanos con otros animales. Actualmente el récord de sitios alternativos de empalme lo tiene el gen Dscam de Drosophila con 38 000 variantes de splicing.
Factores que afectan al splicing alternativo
[editar]Cromatina I
[editar]El contexto cromatínico afecta la tasa de elongación de Polimerasa II, lo que a su vez modifica el splicing alternativo. Se ha encontrado que la despolarización de membrana en células nerviosas afecta el splicing alternativo del pre-mRNA mediante la acetilación intragénica de histonas que relaja la cromatina permitiendo mayor elongación transcripcional.[7]
Cromatina II
[editar]Se ha encontrado también que RNAs pequeños de interferencia (small interfering RNAs, siRNAs) dirigidos contra el intrón río abajo de un exón alternativo afectan el splicing alternativo a través de un mecanismo conocido como silenciamiento génico transcripcional (transcriptional gene silencing, o TGS). Los siRNAs intrónicos gatillan la heterocromatinización en los sitios blanco en el DNA por medio de la dimetilación de la lisina 9 de la histona H3 (H3K9me) y consecuente inhibición de la elongación transcripcional, que a su vez afecta el splicing alternativo.[7] El efecto de los siRNAs intrónicos sobre el splicing alternativo no está relacionado con el silenciamiento génico post-transcripcional. Encontramos que la proteína argonauta AGO1 es necesaria para obtener este efecto sobre la estructura cromatínica. Estamos llevando a cabo un estudio sistemático para identificar en el genoma humano sitios blanco de AGO1 con un rol en el control del splicing alternativo por siRNAs.[7]
Técnicas usadas para su estudio
[editar]- Biología molecular básica (clonado, marcado, hibridación, electroforesis en geles de agarosa y de poliacrilamida, PCR, RT-PCR, PCR en tiempo real)
- Cultivo de células de mamíferos
- Transfecciones de células con DNA y con RNA
- Interferencia por RNA
- Inmunoprecipitación de la cromatina (ChIP)
- Ensayo de accesibilidad de cromatina por MspI
- ChIP-seq
- Western blotting
- Inmunofluorescencia
- Ensayo de protección a la Rnasa (RPA)
- Ensayo de Run-on
- Expresión de genes usando RNA polimerasas mutantes resistentes a alfa-amanitina
- Manipulaciones básicas de Arabidopsis[8]
Mecanismos que utiliza el splicing alternativo
[editar]Se ha determinado en recientes investigaciones que el splicing alternativo en Drosophila determina el sexo del individuo, este mecanismo tiene lugar gracias a una compleja regulación donde intervienen proteínas y muchos factores de los cuales se ha podido resaltar la importancia de la proteína Is.[9]
Partiendo de esta investigación se ha determinado también que en los diferentes sistemas de tejidos existen tipos específicos de splicing alternativo como:
- Elementos exónicos regulatorios y las proteínas que se unen a ellos
- Regulación Negativa en exones
- Elementos intrónicos regulatorios
- Regulación positiva de los intrones
- Regulación negativa de los intrones
- Sistemas multifactoriales de control de splicing
- Proteínas regulatorias de alta eficiencia del tejido
- Disección genétoca de complejos sistemas de regulación[9]
La mayoría de los mecanismos mencionados aún no se han dilucidado a exactitud pero existen ciertas nociones de los mecanismos propuestos, los cuales se expondrán a detalle.
Cuando el pre-mRNA se ha transcrito a partir del ADN, que incluye varios intrones y exones. (En los nematodos, la media es de 4-5 exones e intrones, en la mosca de la fruta Drosophila no puede contener más de 100 intrones y exones en una trascripción de pre-mRNA.) Los exones que deben conservarse en el ARNm se determinan durante el proceso de empalme. La regulación y selección de los sitios de empalme se realizan mediante el activador de splicing trans y las proteínas represoras de splicing, así como elementos que actúan en cis dentro del propio pre-ARNm, tales como potenciadores de empalme exónicos y silenciadores exónicos de splicing.
Elementos intrónicos regulatorios
[editar]
El intrón nuclear eucariótico típico tiene secuencias de consenso que definen regiones importantes. Cada intrón tiene GU en su extremo 5'. Cerca del extremo 3' hay un sitio de la rama. El nucleótido en el punto de ramificación es siempre A; el consenso alrededor de esta secuencia varía algo. En seres humanos, la secuencia de consenso del sitio de la rama es yUnAy. El sitio de ramificación es seguido por una serie de pirimidinas —el tracto polypyrimidine— entonces por AG en el extremo 3 '.[7]
Empalme de mRNA se lleva a cabo por un complejo de ARN y proteína conocida como la spliceosome, que contiene snRNPs U1 designado, U2, U4, U5, U6 y (U3 no está involucrado en mRNA de empalme). U1 se une a la 'GU 5 y U2, con la ayuda de los U2AF factores proteicos, se une a la branchpoint A dentro del sitio de ramificación. El complejo en esta etapa se conoce como el complejo del spliceosome A. La formación del complejo A es usualmente el paso clave para determinar los extremos del intrón a ser empalmados y definir los extremos del exón a retener. (La nomenclatura T se debe a su alto contenido de uridina).[2]
El complejo U4, U5, U6 se une y U6 sustituye a la posición U1. U1 y U4 se van. El complejo restante a continuación, realiza dos de transesterificación reacciones. En el primero de transesterificación, extremo 5' del intrón se escinde del exón aguas arriba y se unió al sitio de la rama A por un 2', 5'- fosfodiéster de ligamiento. En la segunda transesterificación, el extremo 3' del intrón se escinde del exón aguas abajo, y los dos exones están unidos por un enlace fosfodiéster. El intrón se libera entonces en forma de lazo y se degrada.[9]
Regulación positiva de Intrones
[editar]Empalme está regulada por que actúan en trans proteínas (represores y activadores) y correspondientes que actúan en cis sitios de regulación (silenciadores y potenciadores) en el pre-mRNA. Sin embargo, como parte de la complejidad del empalme alternativo, se observa que los efectos de un factor de empalme son frecuentemente dependientes de la posición. Es decir, un factor de empalme, que sirve como un activador de empalme cuando está unido a un elemento potenciador intrónico, puede servir como represor cuando está unido a su elemento de empalme en el contexto de un exón, y viceversa. La estructura secundaria de la transcripción pre-mRNA también juega un papel en la regulación de empalme, como por ejemplo, reuniendo los elementos de corte y empalme o mediante el enmascaramiento de una secuencia que de otro modo servir como un elemento de unión para un factor de empalme. En conjunto, estos elementos forman un "código de empalme" que rige cómo se producirá el corte y empalme bajo diferentes condiciones celulares.[9]
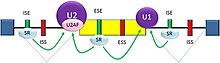
Hay dos tipos principales de cis-actuando elementos de la secuencia de ARN presentes en la pre-ARNm correspondientes y tienen que actúan en trans las proteínas de unión a ARN. Empalme silenciadores son sitios a los que se unen las proteínas represoras de empalme, lo que reduce la probabilidad de que un sitio cercano será utilizado como un nudo de empalme. Estos pueden estar situados en el intrón en sí (empalme silenciadores intrónica, ISS) o en un exón vecino ( silenciadores de empalme exonic, ESS). Varían en secuencia, así como en los tipos de proteínas que se unen a ellos. La mayoría de los represores de empalme son ribonucleoproteínas nucleares heterogéneas (hnRNPs) como hnRNPA1 y proteína de unión del tracto polypyrimidine (PTB). de empalme potenciadores son sitios a los que se unen proteínas activadoras de empalme, el aumento de la probabilidad de que un sitio cercano será utilizado como un nudo de empalme. Estos también pueden ocurrir en los intrones (potenciadores intronic empalme, ISE) o (exón empalme potenciadores exonic, ESE). La mayoría de las proteínas activadoras que se unen a ISEs y ESEs son miembros de la proteína SR familia. Dichas proteínas contienen motivos de reconocimiento de ARN y arginina y dominios ricos en serina (RS).[9]
Regulación negativa de Intrones
[editar]
los determinantes del empalme funcionan de una manera interdependiente que depende del contexto, de modo que las reglas que gobiernan cómo el empalme se regula a partir de un código de empalme. La presencia de un particular, que actúan en cis RNA elemento de secuencia puede aumentar la probabilidad de que un sitio cercano será empalmado en algunos casos, pero disminuir la probabilidad de que en otros casos, dependiendo del contexto. El contexto en el que actúan los elementos de regulación incluye actúa en cis contexto que se establece por la presencia de otras características de secuencia de ARN, y que actúan en trans contexto que se establece por las condiciones celulares. Por ejemplo, algunos actúan en cis elementos de la secuencia de ARN influyen empalme sólo si múltiples elementos están presentes en la misma región a fin de establecer el contexto. Como otro ejemplo, un elemento que actúa en cis puede tener efectos opuestos sobre el empalme, dependiendo de que las proteínas se expresan en la célula (por ejemplo, neuronal versus no neuronal PTB). El significado adaptativo de los silenciadores de empalme y potenciadores es atestiguado por los estudios que muestran que hay una fuerte selección en los genes humanos contra las mutaciones que producen nuevos silenciadores o alterar los potenciadores existentes.[8]
Véase también
[editar]Referencias
[editar]- ↑ Chen; Manley, Mo; James (23 de septiembre de 2009). «Mechanisms of alternative splicing regulation: insights from molecular and genomics approaches.». Nature reviews Molecular cell biology. Consultado el 10 de noviembre de 2009.
- ↑ a b c David Brett; Heike Pospisil; Juan Valcárcel; Jens Reich; Peer Bork (17 de diciembre de 2001). «Alternative splicing and genome complexity». Nature Genetics 30: 29-30. doi:10.1038/ng803.
- ↑ a b Johnson, Castle, Garrett-Engele, Kan, Loerch, Armour, Santos, Schadt, Stoughton, Shoemaker., Jason, John, Philip, Zhengyan, Patrick, Christopher, Ralph, Erick, Roland, Daniel. (19 de diciembre de 2003). «Genome-wide survey of human alternative pre-mRNA splicing with exon junction microarrays.». Science. PMID 14684825. doi:10.1126/science.1090100. Consultado el 19 de diciembre de 2003.
- ↑ Telenti, A., et al. (2016). «Deep sequencing of 10,000 human genomes». Proceedings of the National Academy of Sciences, 201613365.
- ↑ Wang, Cooper., Guey-Shin, Thomas. (8 de octubre de 2007). «Splicing in disease: disruption of the splicing code and the decoding machinery.». Nature Reviews Genetics. PMID 17726481. doi:10.1038/nrg2164. Consultado el 8 de octubre de 2007.
- ↑ a b Zahler., Alan (26 de septiembre de 2005). «8». WormBook: The Online Review of C. elegans Biology (en inglés). ISBN 1551-8507
|isbn=incorrecto (ayuda). - ↑ a b c d Stamm, Stefan (18 de enero de 2008). «Regulation of Alternative Splicing by Reversible Protein Phosphorylation». The Journal of Biological Chemistry. PMID 18024427. doi:10.1074/jbc.R700034200. Consultado el 18 de enero de 2008.
- ↑ a b Shin, Manley., Chanseok, James. (6 de septiembre de 2004). «Cell signalling and the control of pre-mRNA splicing.». Nature Reviews Molecular Cell Biology. PMID 15340380. doi:10.1038/nrm1467. Consultado el 6 de septiembre de 2004.
- ↑ a b c d e Nilsen, Graveley., Timothy, Brenton. (28 de enero de 2010). «Expansion of the eukaryotic proteome by alternative splicing.». Nature. PMID 20110989. doi:10.1038/nature08909. Consultado el 28 de enero de 2010.