GroEL

| GroEL | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Heat Shock Protein Family D (Hsp60) Member 1 | |
| Symbole | HSPD1 | |
| Synonymes | Chaperonine 60 kDa | |
| N° EC | 5.6.1.7 | |
| Homo sapiens | ||
| Locus | 2q33.1 | |
| Masse moléculaire | 61 055 Da[1] | |
| Nombre de résidus | 573 acides aminés[1] | |
| Entrez | 3329 | |
| HUGO | 5261 | |
| OMIM | 118190 | |
| UniProt | P10809 | |
| RefSeq (ARNm) | NM_002156.4, NM_199440.1 | |
| RefSeq (protéine) | NP_002147.2, NP_955472.1 | |
| Ensembl | ENSG00000144381 | |
| PDB | 4PJ1, 6HT7, 6MRC, 6MRD, 7AZP | |
| GENATLAS • GeneTests • GoPubmed • HCOP • H-InvDB • Treefam • Vega | ||
| Liens accessibles depuis GeneCards et HUGO. | ||
| N° EC | EC |
|---|
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
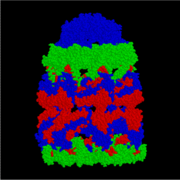
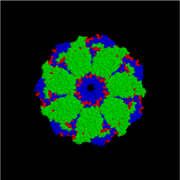
La protéine GroEL appartient à la famille des chaperonines des molécules chaperonnes, et se trouve chez un grand nombre de bactéries[3]. Elle est nécessaire pour le repliement efficace de nombreuses protéines. Afin de fonctionner efficacement, GroEL requiert le complexe protéique couvercle associé GroES. Chez les eucaryotes, les protéines Hsp60 et Hsp10 sont structurellement et fonctionnellement presque identiques à GroEL et GroES, respectivement. Chez l'homme, la protéine GroEL est un dodécamère en forme de ballon de rugby et est codée par le gène HSPD1, situé sur le chromosome 2.
Mécanisme d'action
[modifier | modifier le code]Dans une cellule, le processus de repliement des protéines par médiation GroEL/ES implique de nombreuses séquences de liaisons, encapsulations et libération de protéine substrat.
Les protéines substrats non repliées se lient à une aire hydrophobe sur le bord intérieur de la cavité ouverte du GroEL, formant ainsi un complexe binaire avec la chaperonine. La liaison de protéine substrat de cette façon, en plus de la liaison d'ATP, conduit à une modification conformationnelle qui permet l'association du complexe binaire avec une structure couvercle distincte, GroES. La liaison de GroES avec la cavité ouverte de la chaperonine provoque la rotation des sous-unités individuelles de la chaperonine de manière que le substrat hydrophobe liant soit soustrait à l'intérieur de la cavité, ce qui induit l'éjection de la protéine substrat du bord vers la chambre maintenant hydrophile. L'environnement hydrophile de la chambre favorise l'enfouissement des résidus hydrophobes du substrat, ce qui induit le repliement du substrat. L'hydrolyse de l'ATP et la liaison d'une nouvelle protéine substrat dans la cavité opposée envoient un signal allostérique qui provoque la libération de GroES et d'une protéine encapsulée dans le cytosol.
Une protéine donnée subira plusieurs cycles de repliement, retournant à chaque fois à son état non-replié original, jusqu'à ce que la conformation native ou une structure intermédiaire destinée à atteindre l'état natif soit produite. De manière alternative, le substrat peut être dénaturé lors d'une réaction compétitive, comme un mauvais repliement ou une agrégation avec d'autres protéines mal repliées[4].
Thermodynamique de la réaction
[modifier | modifier le code]La nature contractée de l'intérieur du complexe moléculaire favorise fortement les conformations moléculaires compactes de la protéine substrat. En solution, les interactions apolaires et à longues portées peuvent seulement se produire avec un coût important en entropie. Dans les espaces fermés du complexe GroEL, la perte relative d'entropie est beaucoup plus faible. La méthode de capture tend aussi à concentrer les sites liants apolaires séparément des sites polaires. Lorsque les surfaces apolaires de GroEL sont ôtées, la chance pour qu'un groupe apolaire donné puisse rencontrer un site apolaire intramoléculaire sont bien plus importantes qu'en solution. Les sites hydrophobes se situant à l'extérieur sont rassemblés au sommet du domaine cis et liés entre eux. La géométrie de GroEL impose que les structures polaires soient motrices, et elles enveloppent le cœur apolaire lorsqu'il émerge depuis le côté trans.
Structure
[modifier | modifier le code]GroEL est, structurellement, un tétradécamère à anneaux doubles, les anneaux cis et trans étant constitués de sept sous-unités chacun. L'intérieur de GroEL est hydrophobe, et constitue l'endroit où le repliement des protéines est censé se faire.
- GroEL (côté)
- GroEL (dessus)
- Complexe GroES/GroEL (côté)
- Complexe GroES/GroEL (dessus)
La clé de l'activité de GroEL réside dans la structure du monomère. Le monomère Hsp60 possède trois parties distinctes séparées par deux régions charnières. La région apicale contient un grand nombre de sites liants pour les substrats pour protéines non repliées. De nombreuses protéines globulaires ne se lieront pas à cette région apicale car leurs parties hydrophobes sont internes, loin du milieu aqueux, dans une conformation favorisée par la thermodynamique. En conséquence, ces « sites substrats » ne se lieront qu'avec des protéines qui ne sont pas repliées de manière optimale. La région apicale possède aussi des sites liants pour les monomères Hsp10 de GroES.
Le domaine équatorial possède un emplacement près du point charnière pour lier l'ATP, ainsi que deux points de rattachement pour l'autre moitié de la molécule GroEL. Le reste de la région équatoriale est modérément hydrophile.
L'ajout de l'ATP et de GroES induit un changement radical de la conformation de la région cis. Il est causé par le pliement et la rotation des monomères Hsp60 aux points charnières. La région intermédiaire se replie vers l'intérieur, d'environ 25° par rapport à la charnière la plus basse. Cet effet, multiplié par le pliement coopératif des monomères, accroît le diamètre équatorial de la cage GroEL. Mais le domaine apical pivote de 60 degrés vers l'extérieur par rapport à la charnière supérieure, et aussi de 90 degrés autour de l'axe charnière. Ce mouvement ouvre très largement la cage au-dessus de la région cis, mais supprime l'ensemble des sites liant de l'intérieur de la cage.
Interactions
[modifier | modifier le code]Il a été montré que GroEL interagit avec GroES[5],[6], l'ALDH2[6], la caspase 3[5],[7] et la dihydrofolate réductase[8].
Références
[modifier | modifier le code]- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « GroEL » (voir la liste des auteurs).
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Cecilia Bartolucci, Doriano Lamba, Saulius Grazulis, Elena Manakova et Hermann Heumann, « Crystal Structure of Wild-type Chaperonin GroEL », Journal of Molecular Biology, vol. 354, no 4, , p. 940-951 (PMID 16288915, DOI 10.1016/j.jmb.2005.09.096, lire en ligne)
- (en) Zeilstra-Ryalls J, Fayet O, Georgopoulos C, « The universally conserved GroE (Hsp60) chaperonins », Annu Rev Microbiol., vol. 45, , p. 301 (PMID 1683763, DOI 10.1146/annurev.mi.45.100191.001505).
- (en) Horwich AL, Fenton WA, Chapman E, Farr GW, « Two Families of Chaperonin: Physiology and Mechanism », Annu Rev Cell Dev Biol., vol. 23, , p. 115 (PMID 17489689, DOI 10.1146/annurev.cellbio.23.090506.123555)
- (en) Samali A, Cai J, Zhivotovsky B, Jones D P, Orrenius S, « Presence of a pre-apoptotic complex of pro-caspase-3, Hsp60 and Hsp10 in the mitochondrial fraction of jurkat cells », EMBO J., Angleterre, vol. 18, no 8, , p. 2040–8 (ISSN 0261-4189, PMID 10205158, DOI 10.1093/emboj/18.8.2040)
- (en) Lee Ki-Hwan, Kim Ho-Seung, Jeong Han-Seung, Lee Yong-Sung, « Chaperonin GroESL mediates the protein folding of human liver mitochondrial aldehyde dehydrogenase in Escherichia coli », Biochem. Biophys. Res. Commun., États-Unis, vol. 298, no 2, , p. 216–24 (ISSN 0006-291X, PMID 12387818)
- (en) Xanthoudakis S, Roy S, Rasper D, Hennessey T, Aubin Y, Cassady R, Tawa P, Ruel R, Rosen A, Nicholson D W, « Hsp60 accelerates the maturation of pro-caspase-3 by upstream activator proteases during apoptosis », EMBO J., Angleterre, vol. 18, no 8, , p. 2049–56 (ISSN 0261-4189, PMID 10205159, DOI 10.1093/emboj/18.8.2049)
- (en) Mayhew M, da Silva A C, Martin J, Erdjument-Bromage H, Tempst P, Hartl F U, « Protein folding in the central cavity of the GroEL-GroES chaperonin complex », Nature, Angleterre, vol. 379, no 6564, , p. 420–6 (ISSN 0028-0836, PMID 8559246, DOI 10.1038/379420a0)
Voir aussi
[modifier | modifier le code]Bibliographie
[modifier | modifier le code]- (en) Tabibzadeh S, Broome J, « Heat shock proteins in human endometrium throughout the menstrual cycle », Infectious diseases in obstetrics and gynecology, vol. 7, nos 1-2, , p. 5–9 (PMID 10231001, DOI 10.1002/(SICI)1098-0997(1999)7:1/2<5::AID-IDOG2>3.0.CO;2-Y)
- (en) Schäfer C, Williams JA, « Stress kinases and heat shock proteins in the pancreas: possible roles in normal function and disease », J. Gastroenterol., vol. 35, no 1, , p. 1–9 (PMID 10632533)
- (en) Moseley P, « Stress proteins and the immune response », Immunopharmacology, vol. 48, no 3, , p. 299–302 (PMID 10960671, DOI 10.1016/S0162-3109(00)00227-7)
- (en) Liu Y, Steinacker JM, « Changes in skeletal muscle heat shock proteins: pathological significance », Front. Biosci., vol. 6, , D12–25 (PMID 11145923, DOI 10.2741/Liu)
- (en) Van Maele B, Debyser Z, « HIV-1 integration: an interplay between HIV-1 integrase, cellular and viral proteins », AIDS reviews, vol. 7, no 1, , p. 26–43 (PMID 15875659)
- (en) Hochstrasser DF, Frutiger S, Paquet N, et al., « Human liver protein map: a reference database established by microsequencing and gel comparison », Electrophoresis, vol. 13, no 12, , p. 992–1001 (PMID 1286669, DOI 10.1002/elps.11501301201)
- (en) Ikawa S, Weinberg RA, « An interaction between p21ras and heat shock protein hsp60, a chaperonin », Proc. Natl. Acad. Sci. U.S.A., vol. 89, no 6, , p. 2012–6 (PMID 1347942, DOI 10.1073/pnas.89.6.2012)
- (en) Brudzynski K, Martinez V, Gupta RS, « Immunocytochemical localization of heat-shock protein 60-related protein in beta-cell secretory granules and its altered distribution in non-obese diabetic mice », Diabetologia, vol. 35, no 4, , p. 316–24 (PMID 1516759, DOI 10.1007/BF00401198)
- (en) Dawson SJ, White LA, « Treatment of Haemophilus aphrophilus endocarditis with ciprofloxacin », J. Infect., vol. 24, no 3, , p. 317–20 (PMID 1602151, DOI 10.1016/S0163-4453(05)80037-4)
- (en) Singh B, Patel HV, Ridley RG, et al., « Mitochondrial import of the human chaperonin (HSP60) protein », Biochem. Biophys. Res. Commun., vol. 169, no 2, , p. 391–6 (PMID 1972619, DOI 10.1016/0006-291X(90)90344-M)
- (en) Venner TJ, Singh B, Gupta RS, « Nucleotide sequences and novel structural features of human and Chinese hamster hsp60 (chaperonin) gene families », DNA Cell Biol., vol. 9, no 8, , p. 545–52 (PMID 1980192, DOI 10.1089/dna.1990.9.545)
- (en) Ward LD, Hong J, Whitehead RH, Simpson RJ, « Development of a database of amino acid sequences for human colon carcinoma proteins separated by two-dimensional polyacrylamide gel electrophoresis », Electrophoresis, vol. 11, no 10, , p. 883–91 (PMID 2079031, DOI 10.1002/elps.1150111019)
- (en) Jindal S, Dudani AK, Singh B, et al., « Primary structure of a human mitochondrial protein homologous to the bacterial and plant chaperonins and to the 65-kilodalton mycobacterial antigen », Mol. Cell. Biol., vol. 9, no 5, , p. 2279–83 (PMID 2568584)
- (en) Waldinger D, Eckerskorn C, Lottspeich F, Cleve H, « Amino-acid sequence homology of a polymorphic cellular protein from human lymphocytes and the chaperonins from Escherichia coli (groEL) and chloroplasts (Rubisco-binding protein) », Biol. Chem. Hoppe-Seyler, vol. 369, no 10, , p. 1185–9 (PMID 2907406)
- (en) Kreisel W, Hildebrandt H, Schiltz E, et al., « Immuno-gold electron microscopical detection of heat shock protein 60 (hsp60) in mitochondria of rat hepatocytes and myocardiocytes », Acta Histochem., vol. 96, no 1, , p. 51–62 (PMID 7518175)
- (en) Corbett JM, Wheeler CH, Baker CS, et al., « The human myocardial two-dimensional gel protein database: update 1994 », Electrophoresis, vol. 15, no 11, , p. 1459–65 (PMID 7895732, DOI 10.1002/elps.11501501209)
- (en) Baca-Estrada ME, Gupta RS, Stead RH, Croitoru K, « Intestinal expression and cellular immune responses to human heat-shock protein 60 in Crohn's disease », Dig. Dis. Sci., vol. 39, no 3, , p. 498–506 (PMID 7907543, DOI 10.1007/BF02088334)
- (en) Vélez-Granell CS, Arias AE, Torres-Ruíz JA, Bendayan M, « Molecular chaperones in pancreatic tissue: the presence of cpn10, cpn60 and hsp70 in distinct compartments along the secretory pathway of the acinar cells », J. Cell. Sci., vol. 107 ( Pt 3), , p. 539–49 (PMID 7911805)
- (en) Mayhew M, da Silva AC, Martin J, et al., « Protein folding in the central cavity of the GroEL-GroES chaperonin complex », Nature, vol. 379, no 6564, , p. 420–6 (PMID 8559246, DOI 10.1038/379420a0)
- (en) Tabibzadeh S, Kong QF, Satyaswaroop PG, Babaknia A, « Heat shock proteins in human endometrium throughout the menstrual cycle », Hum. Reprod., vol. 11, no 3, , p. 633–40 (PMID 8671282)
- (en) P. D. Kiser, G. H. Lorimer et K. Palczewski, « Use of thallium to identify monovalent cation binding sites in GroEL », Acta Crystallographica Section F: Structural Biology and Crystallization Communications, vol. 65, no 10, , p. 967–971 (ISSN 1744-3091, DOI 10.1107/S1744309109032928, lire en ligne, consulté le )
Articles connexes
[modifier | modifier le code]Liens externes
[modifier | modifier le code]- (es) « Palaeos Bacteria: Pieces: GroEL » (pas de droits réservés)
- Ressource relative à la santé :