タンパク質を構成しないアミノ酸
 ウィキペディアから無料の百科事典
ウィキペディアから無料の百科事典

タンパク質を構成しないアミノ酸(タンパクしつをこうせいしないアミノさん、英: Non-proteinogenic amino acids)は、天然においては、生物の遺伝コード(コドン)に見られないアミノ酸である。タンパク質を組み立てるための翻訳装置では22個(真核生物では21個)のアミノ酸しか用いられないが、天然のタンパク質から得られるアミノ酸としては140以上が知られており、天然で生成または実験室で合成されるアミノ酸としては、数千が知られている[1]。
タンパク質を構成しないアミノ酸の多くは、以下の点で注目すべきである。
- 生合成の中間体である
- タンパク質中で翻訳後に形成される
- 生理学的役割を持つ(細菌の細胞壁(ペプチドグリカン)、神経伝達物質、毒素)
- 薬理作用を持つ天然または人工の化合物である
- 隕石中や生物誕生前の環境にも存在する(ユーリー-ミラーの実験)
否定による定義[編集]

官能基としてアミンとカルボン酸を持つ全ての有機化合物がアミノ酸である。タンパク質を構成するアミノ酸はその中の一部であり、中央の炭素原子(α-または2-)が左旋性でアミノ基、カルボキシル基、側鎖、α-水素原子を持つ。例外は、アキラルであるグリシンと、アミノ基が第二級アミンでありイミノ基を持たないもののしばしばイミノ酸とも呼ばれるプロリンである。
遺伝コードは、翻訳によってタンパク質に取り込まれる20個の標準アミノ酸をコードしている。しかし、タンパク質を構成するアミノ酸としてはさらに2つ、セレノシステインとピロリシンがある。この2つにはコドンは割り当てられていないが、特殊な配列が存在する場合に終止コドンの位置に加えられる。即ち、セレノシステイン挿入配列 (SecIS) があった場合にUGAコドンがセレノシステインに翻訳され[2]、PYLIS配列があった場合にUAGコドンがピロリシンに翻訳される[3]。これら以外の全てのアミノ酸が「タンパク質を構成しないアミノ酸」である。
- セレノシステインは、β-炭素にセレノール基を含む。
- ピロリシンは、リシンのε-アミノ基にカルボキシル化ピロリン環が付いたものである。
アミノ酸のグループとしては、以下のようなものがある[4]。
- 20の標準アミノ酸
- 22のタンパク質を構成するアミノ酸
- 80以上の高濃度で非生物的に合成されるアミノ酸
- 900程度の天然経路で合成されるアミノ酸
- 118以上のタンパク質に組み込まれる人工アミノ酸
これらのグループには重複はあるが、同一のものではない。22のタンパク質を構成するアミノ酸は全て生物等によって生合成されるが、発生源はそれだけではなく、非生物的(生物誕生前の環境や隕石中)にも生じうる。ノルロイシン等のいくつかの天然アミノ酸は、タンパク質合成プロセスがそれほど厳密ではないために、誤ってタンパク質に取り入れられることがある。オルニチン等の多くのアミノ酸は、生合成で生み出される代謝中間体であるが、タンパク質に取り入れられることはない。タンパク質中でのアミノ酸残基の翻訳後修飾により、タンパク質の一部ではあるがタンパク質を構成するアミノ酸ではないアミノ酸が多く形成される。α-メチルノルバリン等の他のアミノ酸は、非生物的な環境でのみ見られる。エンジニアリングされた系において、30以上の非天然アミノ酸が翻訳によってタンパク質に組み込まれるが、これは生合成的ではない[4]。
命名法[編集]
カルボキシル基中のものも含め、分子を構成する各炭素原子に順番に番号を割り振ることによって有機分子中の様々な炭素原子を区別するためのIUPACナンバリング体系に加え、アミノ酸の側鎖上の炭素原子にギリシャ文字を割り振ることができる。この際、α炭素は、カルボキシル基と側鎖、さらにα-アミノ酸の場合はアミノ基を持つ中央のキラル炭素となり、カルボキシル基の炭素は数えない[5](したがって、タンパク質を構成しないα-アミノ酸の多くの名前は、「2-アミノ」で始まる)。
L-α-アミノ酸以外の天然アミノ酸[編集]
天然に存在するアミノ酸のほとんどは、L型のα-アミノ酸であるが、例外も存在する。
α-アミノ酸以外[編集]

生物中にもいくつかの非α-アミノ酸が存在する。これらの構造では、アミン基は、アミノ酸分子の端にあるカルボキシル基から遠い位置に置かれる。β-アミノ酸は2番目、γ-アミノ酸は3番目の炭素上にアミン基が置かれる。例えば、β-アラニン、γ-アミノ酪酸、δ-アミノレブリン酸等がある。
- δ-アミノレブリン酸は、テトラピロール(ヘム、クロロフィル、コバラミン等)生合成の中間体である。
かつては、β-アミノ酸の二次構造が有害であると考えられたが[7]、間違いであることが明らかとなった[8]。
D-アミノ酸[編集]
いくつかのアミノ酸はキラリティーが反対で、通常のリボソーム転写/翻訳はされない。細菌の細胞壁の大部分は、アミノ糖が短いオリゴペプチドで架橋されたペプチドグリカンでできている。オリゴペプチドは非リボソーム的に合成され、D-アラニンやD-グルタミン酸等の特殊なアミノ酸を含む。さらに前者はalr遺伝子またはホモログのdadX遺伝子でコードされるピリドキサールリン酸結合酵素により、後者は補因子に依存しない酵素 (murI) によってラセミ化されている。テルモトガ属はD-リシン、バンコマイシン耐性細菌はD-セリン(vanT遺伝子)を持つ[9][10]。
動物では、いくつかのD-アミノ酸は、神経伝達物質である。
水素を欠くα-炭素[編集]
タンパク質を構成するアミノ酸は全て、α炭素上に少なくとも1つの水素原子を持つ。グリシンは2つの水素原子を持ち、その他は全て1つの水素原子と1つの側鎖を持つ。水素原子をメチル基で置換すると、タンパク質の主鎖が歪む[7]。
菌類の一部では、抗菌活性を持つペプチドの前駆体として2-アミノイソ酪酸を生産する[11]。これはアラニンに似ているが、α炭素上の水素の代わりにメチル基を持つため、アキラルである。α水素を持たないアラニン様物質としては、他にメチレン側鎖を持つデヒドロアラニンがある。これは、天然に存在するいくつかのデヒドロアミノ酸のうちの1つである。
- アラニン
- アミノイソ酪酸
- デヒドロアラニン
双子アミノ酸の立体中心[編集]
L-α-アミノ酸の一部では、2つの末端のどちらがα炭素であるかについては曖昧である。タンパク質中では、システイン残基は他のシステイン残基とジスルフィド結合を形成して、タンパク質を架橋することができる。2つの架橋したシステインは、シスチン分子を形成する。システインとメチオニンは、一般的には直接のスルフリル化で形成されるが、いくつかの種はトランススルフレーション経路で形成され、ここでは活性化されたホモセリンまたはセリンがシステインまたはホモシステインと融合してシスタチオニンを形成する。似た化合物にランチオニンがあり、2つのアラニン分子がチオエステル結合で結合したもので、様々な生物で見られる。同様に、ジリンマメの毒であるジェンコル酸では、2つのシステインがメチレン基で繋がっている。ジアミノピメリン酸は、ペプチドグリカンの架橋や脱炭酸によりリシンの前駆体として用いられる。
- シスチン
- シスタチオニン
- ランチオニン
- ジエンコル酸
- ジアミノピメリン酸
生物誕生前のアミノ酸と代替生化学[編集]
隕石中やユーリー-ミラーの実験等の生物誕生以前の環境を再現する実験では、20種類よりも多くのアミノ酸が見られ、その中のいくつかは標準よりも高い濃度となった。ここから、宇宙のどこかでアミノ酸を基盤とする生命が全く別に現れたとしても、共通するアミノ酸は高々75%に過ぎないことが予測される[7]。最も顕著なのは、アミノ酪酸が欠けていることである。
| グリシンに対する割合 (%) | ||
|---|---|---|
| 分子 | 放電による生成割合 | マーチソン隕石内の割合 |
| グリシン | 100 | 100 |
| アラニン | 180 | 36 |
| α-アミノ-n-酪酸 | 61 | 19 |
| ノルバリン | 14 | 14 |
| バリン | 4.4 | |
| ノルロイシン | 1.4 | |
| ロイシン | 2.6 | |
| イソロイシン | 1.1 | |
| アロイソロイシン | 1.2 | |
| tert-ロイシン | < 0.005 | |
| α-アミノ-n-ヘプタン酸 | 0.3 | |
| プロリン | 0.3 | 22 |
| ピペコリン酸 | 0.01 | 11 |
| α,β-ジアミノプロピオン酸 | 1.5 | |
| α,γ-ジアミノ酪酸 | 7.6 | |
| オルニチン | < 0.01 | |
| リシン | < 0.01 | |
| アスパラギン酸 | 7.7 | 13 |
| グルタミン酸 | 1.7 | 20 |
| セリン | 1.1 | |
| トレオニン | 0.2 | |
| アロトレオニン | 0.2 | |
| メチオニン | 0.1 | |
| ホモシステイン | 0.5 | |
| ホモセリン | 0.5 | |
| β-アラニン | 4.3 | 10 |
| β-アミノ-n-酪酸 | 0.1 | 5 |
| β-アミノイソ酪酸 | 0.5 | 7 |
| γ-アミノ酪酸 | 0.5 | 7 |
| α-アミノイソ酪酸 | 7 | 33 |
| イソバリン | 1 | 11 |
| サルコシン | 12.5 | 7 |
| N-エチルグリシン | 6.8 | 6 |
| N-プロピルグリシン | 0.5 | |
| N-イソプロピルグリシン | 0.5 | |
| N-メチルアラニン | 3.4 | 3 |
| N-エチルアラニン | < 0.05 | |
| N-メチル-β-アラニン | 1.0 | |
| N-エチル-β-アラニン | < 0.05 | |
| イソセリン | 1.2 | |
| α-ヒドロキシ-γ-アミノ酪酸 | 17 | |
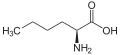
直鎖状側鎖[編集]
遺伝コードについては、進化の過程で遺伝コードが成立する際に偶然決まり、そのまま現在まで凍結されたものだとする偶然凍結説が提唱されており、20種類の標準アミノ酸の中で直鎖状の側鎖を持つものがアラニンのみである理由は、単純にバリン、ロイシン、イソロイシンの冗長さのためかもしれない[7]。しかし、直鎖状側鎖を持つアミノ酸は、より安定なαヘリックスを形成すると報告されている[12]。
- グリシン(水素側鎖)
- アラニン(メチル側鎖)
- α-アミノ酪酸(エチル側鎖)
- ノルバリン(n-プロピル側鎖)
- ノルロイシン(n-ブチル側鎖)
- ホモノルロイシン(n-ペンチル側鎖)
カルコゲン[編集]
セリン、ホモセリン、O-メチルホモセリン、O-エチルホモセリンは、それぞれ、ヒドロキシメチル基、ヒドロキシエチル基、O-メチルヒドロキシメチル基、O-メチルヒドロキシエチル基を側鎖に持つ。一方、システイン、ホモシステイン、メチオニン、エチオニンは、それぞれのチオール置換体である。セレノール置換体は、セレノシステイン、セレノホモシステイン、セレノメチオニン、セレノエチオニンである。Aspergillus fumigatus、Aspergillus terreus、Penicillium chrysogenum等のいくつかの種は、硫黄のない環境では、テルロシステイン、テルロメチオニンを作ってタンパク質に取り込むことができる[13]。
ヒドロキシル側鎖を持つヒドロキシグリシンは、非常に不安定である。
拡張遺伝コード[編集]
役割[編集]
細胞内、特に独立栄養生物の細胞内では、タンパク質を構成しないアミノ酸のいくつかは代謝中間体として見られる。しかし、多くのアミノ酸はケト酸として作られ、最後の段階でアミノ化されるため、タンパク質を構成しないアミノ酸が代謝中間体になることはかなり少ない。
オルニチンとシトルリンは、アミノ酸異化の一部分である尿素回路で生成される[14]。
一次代謝に加え、タンパク質を構成しないアミノ酸のいくつかは、小分子や非リボソームペプチドを作るための二次代謝の前駆体や最終生成物となる。
翻訳後のタンパク質への取込み[編集]
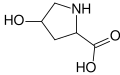
タンパク質を構成するアミノ酸のように遺伝コードでコードされていないが、非標準アミノ酸のいくつかはタンパク質中で見られる。これらは、タンパク質中の標準アミノ酸の側鎖が翻訳後修飾されたものである。このような翻訳後修飾は、しばしばタンパク質の機能や制御に必要なものである。例えば、グルタミン酸のカルボキシル化物であるγ-カルボキシグルタミン酸は、カルシウムカチオンとより結合しやすく[15]、プロリンのヒドロキシル化物であるヒドロキシプロリンは、結合組織(コラーゲン)の維持に不可欠である[16]。他の例として、真核生物の翻訳開始因子EIF5Aに含まれるヒプシンは、リシン残基を修飾したものである[17]。このような修飾は、タンパク質の局在を決めることもある。例えば、長い疎水性の官能基が付加すると、タンパク質はリン脂質膜に結合しやすくなる[18]。
- グルタミン酸は1つのγ-カルボキシル基を持つが、カルボキシグルタミン酸は2つ持つ。
- ヒドロキシプロリンは、プロリンの4番炭素にヒドロキシル基が付いたイミノ酸である。
- ヒプシンは、リシンのε-アミノ基に4-アミノブチル基が付いたものである。
おそらく誤取込みによって、タンパク質中にアミノマロン酸が含まれることもあるという予備的証拠も得られている[19][20]。
毒性アナログ[編集]
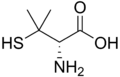
タンパク質を構成しないアミノ酸のうち、チアリシン等は、タンパク質を構成するアミノ酸の特定の性質を模倣するため、毒性を持つ。またキスカル酸、カナバニン、アゼチジン-2-カルボン酸等は、神経伝達物質となるアミノ酸を模倣するため、神経毒となる[21]。セファロスポリンCはホモグルタミン酸骨格がセファロスポリン基でアミド化されている[22]。D-ペニシラミンは、作用機構が未知の治療薬である。
- チアリシン
- キスカル酸
- カナバニン
- アゼチジン-2-カルボン酸
- セファロスポリンC
- ペニシラミン
天然に生成するシアノトキシンもタンパク質を構成しないアミノ酸を含んでいる。例えば、ミクロシスチンやノジュラリンは、どちらもβ-アミノ酸であるADDAに由来する。
非アミノ酸[編集]
タウリンは、アミノスルホン酸でありアミノ酸ではないが、ネコ等の特定の生物では栄養要求を満たすのに必要な量がビタミンよりも必須アミノ酸に近い。
オスモライトであるサルコシンやトリメチルグリシンはアミノ酸に由来するが、それぞれ第2級アミン、第4級アミンを持つ。
出典[編集]
- ^ Ambrogelly, A.; Palioura, S.; Soll, D. (2007). “Natural expansion of the genetic code”. Nature Chemical Biology 3 (1): 29-35. doi:10.1038/nchembio847. PMID 17173027.
- ^ Bock, A.; Forchhammer, K.; Heider, J.; Baron, C. (1991). “Selenoprotein synthesis: An expansion of the genetic code”. Trends in Biochemical Sciences 16 (12): 463-467. doi:10.1016/0968-0004(91)90180-4. PMID 1838215.
- ^ Theobald-Dietrich, A.; Giege, R.; Rudinger-Thirion, J. L. (2005). “Evidence for the existence in mRNAs of a hairpin element responsible for ribosome dependent pyrrolysine insertion into proteins”. Biochimie 87 (9-10): 813-817. doi:10.1016/j.biochi.2005.03.006. PMID 16164991.
- ^ a b Lu, Y.; Freeland, S. (2006). “On the evolution of the standard amino-acid alphabet”. Genome Biology 7 (1): 102. doi:10.1186/gb-2006-7-1-102. PMC 1431706. PMID 16515719.
- ^ Voet, D.; Voet, J. G. (2004). Biochemistry (3rd ed.). John Wiley & Sons. ISBN 978-0471193500
- ^ Chakauya, E.; Coxon, K. M.; Ottenhof, H. H.; Whitney, H. M.; Blundell, T. L.; Abell, C.; Smith, A. G. (2005). “Pantothenate biosynthesis in higher plants”. Biochemical Society Transactions 33 (4): 743-746. doi:10.1042/BST0330743. PMID 16042590.
- ^ a b c d Weber, A. L.; Miller, S. L. (1981). “Reasons for the occurrence of the twenty coded protein amino acids”. Journal of Molecular Evolution 17 (5): 273–284. Bibcode: 1981JMolE..17..273W. doi:10.1007/BF01795749. PMID 7277510.
- ^ Koyack, M. J.; Cheng, R. P. (2006). “Design and Synthesis of β-Peptides With Biological Activity”. Protein Design. Methods in Molecular Biology. 340. pp. 95-109. doi:10.1385/1-59745-116-9:95. ISBN 978-1-59745-116-1. PMID 16957334
- ^ Boniface, A.; Parquet, C.; Arthur, M.; Mengin-Lecreulx, D.; Blanot, D. (2009). “The Elucidation of the Structure of Thermotoga maritima Peptidoglycan Reveals Two Novel Types of Cross-link”. Journal of Biological Chemistry 284 (33): 21856-21862. doi:10.1074/jbc.M109.034363. PMC 2755910. PMID 19542229.
- ^ Arias, C. A.; Martin-Martinez, M.; Blundell, T. L.; Arthur, M.; Courvalin, P.; Reynolds, P. E. (1999). “Characterization and modelling of VanT: A novel, membrane-bound, serine racemase from vancomycin-resistant Enterococcus gallinarum BM4174”. Molecular Microbiology 31 (6): 1653-1664. doi:10.1046/j.1365-2958.1999.01294.x. PMID 10209740.
- ^ Gao, X.; Chooi, Y. H.; Ames, B. D.; Wang, P.; Walsh, C. T.; Tang, Y. (2011). “Fungal Indole Alkaloid Biosynthesis: Genetic and Biochemical Investigation of the Tryptoquialanine Pathway inPenicillium aethiopicum”. Journal of the American Chemical Society 133 (8): 2729-2741. doi:10.1021/ja1101085. PMC 3045477. PMID 21299212.
- ^ Padmanabhan, S.; Baldwin, R. L. (1991). “Straight-chain non-polar amino acids are good helix-formers in water”. Journal of Molecular Biology 219 (2): 135-137. doi:10.1016/0022-2836(91)90553-I. PMID 2038048.
- ^ Ramadan, S. E.; Razak, A. A.; Ragab, A. M.; El-Meleigy, M. (1989). “Incorporation of tellurium into amino acids and proteins in a tellurium-tolerant fungi”. Biological Trace Element Research 20 (3): 225-232. doi:10.1007/BF02917437. PMID 2484755.
- ^ Curis, E.; Nicolis, I.; Moinard, C.; Osowska, S.; Zerrouk, N.; Benazeth, S.; Cynober, L. (2005). “Almost all about citrulline in mammals”. Amino Acids 29 (3): 177-205. doi:10.1007/s00726-005-0235-4. PMID 16082501.
- ^ Vermeer, C. (1990). “Gamma-carboxyglutamate-containing proteins and the vitamin K-dependent carboxylase”. The Biochemical Journal 266 (3): 625-636. doi:10.1042/bj2660625. PMC 1131186. PMID 2183788.
- ^ Bhattacharjee, A; Bansal, M (2005). “Collagen structure: The Madras triple helix and the current scenario”. IUBMB Life (International Union of Biochemistry and Molecular Biology: Life) 57 (3): 161-72. doi:10.1080/15216540500090710. PMID 16036578.
- ^ Park, M. H. (2006). “The post-translational synthesis of a polyamine-derived amino acid, hypusine, in the eukaryotic translation initiation factor 5A (eIF5A)”. Journal of Biochemistry 139 (2): 161-9. doi:10.1093/jb/mvj034. PMC 2494880. PMID 16452303.
- ^ Blenis, J; Resh, M. D. (1993). “Subcellular localization specified by protein acylation and phosphorylation”. Current Opinion in Cell Biology 5 (6): 984-9. doi:10.1016/0955-0674(93)90081-z. PMID 8129952.
- ^ Copley, S. D.; Frank, E.; Kirsch, W. M.; Koch, T. H. (1992). “Detection and possible origins of aminomalonic acid in protein hydrolysates”. Analytical Biochemistry 201 (1): 152-157. doi:10.1016/0003-2697(92)90188-D. PMID 1621954.
- ^ Van Buskirk, J. J.; Kirsch, W. M.; Kleyer, D. L.; Barkley, R. M.; Koch, T. H. (1984). “Aminomalonic acid: Identification in Escherichia coli and atherosclerotic plaque”. Proceedings of the National Academy of Sciences of the United States of America 81 (3): 722-725. doi:10.1073/pnas.81.3.722. PMC 344907. PMID 6366787.
- ^ Dasuri, K.; Ebenezer, P. J.; Uranga, R. M.; Gavilan, E.; Zhang, L.; Fernandez-Kim, S. O. K.; Bruce-Keller, A. J.; Keller, J. N. (2011). “Amino acid analog toxicity in primary rat neuronal and astrocyte cultures: Implications for protein misfolding and TDP-43 regulation”. Journal of Neuroscience Research 89 (9): 1471-1477. doi:10.1002/jnr.22677. PMC 3175609. PMID 21608013.
- ^ Trown, P. W.; Smith, B.; Abraham, E. P. (1963). “Biosynthesis of cephalosporin C from amino acids”. The Biochemical Journal 86 (2): 284-291. doi:10.1042/bj0860284. PMC 1201751. PMID 13994319.

![β-アラニンはアスパラギン酸-1-デカルボキシラーゼによって作られ、補酵素A[6]やペプチドのカルノシン、アンセリンの前駆体となる。](http://upload.wikimedia.org/wikipedia/commons/thumb/3/35/Beta-alanine_structure.svg/120px-Beta-alanine_structure.svg.png)