ヒストンH1
 ウィキペディアから無料の百科事典
ウィキペディアから無料の百科事典
| linker histone H1 and H5 family | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| 識別子 | |||||||||||
| 略号 | Linker_histone | ||||||||||
| Pfam | PF00538 | ||||||||||
| InterPro | IPR005818 | ||||||||||
| SMART | SM00526 | ||||||||||
| SCOP | 1hst | ||||||||||
| SUPERFAMILY | 1hst | ||||||||||
| |||||||||||
ヒストンH1(英: histone H1)は、真核生物細胞のクロマチンの構成要素となる5つの主要なヒストンタンパク質ファミリーの1つである。ヒストンH1は高度に保存されているものの、生物種間の配列の多様性はヒストンの中では最も高い。
構造[編集]
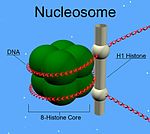
後生動物のH1タンパク質は、中央部の球状の"winged helix"ドメインと長いC末端テール、短いN末端テールという特徴を持つ。H1はヌクレオソームの数珠玉状の"beads on a string"構造から高次構造へのパッキングに関与しているが、その詳細は未解明である[1]。原生生物や細菌に存在するH1はヌクレオタンパク質HC1/HC2 (Pfam PF07382)として知られ、中央部のドメインとN末端テールを欠く[2]。
H1はコアヒストンと比較して保存性が低い。球状ドメインはH1の最も保存性が高い領域である[3]。
機能[編集]
他のヒストンとは異なり、H1はヌクレオソームの「ビーズ」を構成しない。H1はビーズ構造上に位置し、DNAがヌクレオソームの周囲を巻くよう適切な位置に維持している。ヌクレオソームのビーズは4種類のヒストン2分子ずつから構成されるが、H1は1分子である。ヌクレオソームへの結合に加えて、H1タンパク質はヌクレオソーム間の「リンカーDNA」(約20–80ヌクレオチドの長さ)に結合し、ジグザグ型の30 nmクロマチン繊維の安定化を助けている[4]。ヒストンH1に関しては精製されたクロマチン繊維を用いて多くの研究がなされている。天然のクロマチンや再構成されたクロマチンからリンカーヒストンを除去すると、低浸透圧条件下で30 nm繊維からbeads-on-a-string型ヌクレオソームへ高次構造の解消が促進される[5][6][7]。
H1はリンカーDNAの露出部分を短くしてソレノイド状のクロマチン繊維の形成を促進しているのか、それともリンカーの長さには影響を与えることなく単に隣接するヌクレオソームの角度の変化を促進しているだけであるのかは明確ではない[8]。しかしながら、リンカーヒストンは強力な601ヌクレオソームポジショニングエレメントからなる合成DNAを用いてin vitroで再構成されたクロマチン繊維の凝縮を駆動することが示されている[9]。ヌクレアーゼ分解実験やDNAフットプリンティング実験からは、ヒストンH1の球状ドメインはヌクレオソームのダイアド(dyad)部分の近傍に位置していることが示唆されており、ヒストンH1の存在によってさらに約15–30塩基対のDNAが保護される[10][11][12][13]。また、再構成クロマチンを用いた実験では、H1の存在下ではダイアド部分には特徴的なDNAのステムモチーフが生じることが示されている[14]。H1の機能に関しては見解の相違が存在するものの、一般的に受け入れられているモデルは、H1の球状ドメインはヌクレオソームへ入るDNAと出るDNAを架橋して閉じ、テール部分はリンカーDNAに結合してその負電荷を中和する、というものである[8][12]。
H1の機能に関する実験の多くは低塩条件下で精製されプロセシングされたクロマチンを用いて行われているが、in vivoでのH1の役割に関してはさらに不明な点が多い。細胞を用いた研究では、H1の過剰発現は核の形態とクロマチン構造の異常を引き起こすこと、H1は遺伝子によって転写の正の調節因子としても負の調節因子としても作用しうることが示されている[15][16][17]。ツメガエルの卵抽出液では、リンカーヒストンの除去によって分裂期染色体の長さは約2倍となり、過剰発現では染色体は分離不可能な塊へ過剰凝縮される[18][19]。多細胞生物ではH1は複数のアイソフォームがいくつかの遺伝子クラスターとして存在するため、in vivoでのH1の完全なノックアウトは行われていないが、テトラヒメナ、シロイヌナズナ、線虫、ショウジョウバエ、マウスを用いてさまざまなアイソフォームをさまざまな程度で除去する実験が行われており、核の形態やクロマチン構造、DNAメチル化、特定の遺伝子の発現などに生物種特異的なさまざまな欠陥が生じることが示されている[20][21][22]。
ダイナミクス[編集]
核内のヒストンH1の大部分はクロマチンに結合している一方で、H1分子はかなり高率でクロマチン間を移行することが知られている[23][24]。
こうした動的なタンパク質がどのようにクロマチンの構造的構成要素となっているのかを理解するのは難しいが、核内での定常状態の平衡はH1とクロマチンの結合がかなり有利であることが示唆されている。このことは、そのダイナミクスにもかかわらず、どの時点においても大多数のH1はクロマチンに結合した状態であることを意味している[25]。H1はDNAに力がかかっている状態やクロマチンの組み立て時において、DNAを圧縮して安定化することが示されており、H1の動的な結合はヌクレオソームを除去する必要がある状況下でDNAを保護していることが示唆される[26]。
クロマチン上でのヒストンH1の動的な交換には細胞質の因子が必要であるようであるが、具体的には未同定である[27]。H1のダイナミクスの一部はO-グリコシル化とリン酸化によって媒介されている可能性がある。H1のO-グリコシル化はクロマチンの凝縮を促進する可能性がある。間期の間のH1のリン酸化はクロマチンに対する親和性を低下させることが示されており、クロマチンの脱凝縮と活発な転写を促進している可能性がある。しかしながら、有糸分裂期のリン酸化はH1の染色体に対する親和性を増大させ、染色体凝縮を促進することが示されている[19]。
アイソフォーム[編集]
動物のH1ファミリーには複数のH1のアイソフォームが存在し、それらは生体内で異なる組織と発生段階で発現しているものや、重複した領域で発現しているものもある。こうした複数のアイソフォームが存在する理由は明らかではないが、ウニからヒトまで進化的に保存されていることやアイソフォーム間のアミノ酸配列の大きな差異は、これらが機能的に等価なものではないことを示唆している[3][28][29]。1つのアイソフォームはヒストンH5と呼ばれ、このアイソフォームは鳥類の赤血球にのみ見られる。他のアイソフォームとしては卵母細胞/受精卵型アイソフォームH1M(B4またはH1fooとしても知られる)があり、ウニ、カエル、マウス、そしてヒトにも存在し、胚では体細胞型アイソフォームH1A–Eや、H5に似たH1.0アイソフォームへと置き換えられる[3][30][31][32]。H1Mは体細胞型アイソフォームよりも多くの負電荷を持つにもかかわらず、ツメガエルの卵抽出液中の分裂期染色体に対してより高い親和性で結合する[19]。
翻訳後修飾[編集]
他のヒストンと同様、ヒストンH1ファミリーも広範囲にわたって翻訳後修飾を受ける。セリンとスレオニンのリン酸化、リジンのアセチル化、メチル化、ユビキチン化などが含まれる[33]。こうした翻訳後修飾はさまざまな機能を持つが、他のヒストンの翻訳後修飾を比較して研究は進んでいない。
出典[編集]
- ^ “Crystal structure of globular domain of histone H5 and its implications for nucleosome binding”. Nature 362 (6417): 219–23. (March 1993). Bibcode: 1993Natur.362..219R. doi:10.1038/362219a0. PMID 8384699.
- ^ “Origin of H1 linker histones”. FASEB Journal 15 (1): 34–42. (January 2001). doi:10.1096/fj.00-0237rev. PMID 11149891.
- ^ a b c “The histone H1 family: specific members, specific functions?”. Biological Chemistry 389 (4): 333–43. (April 2008). doi:10.1515/BC.2008.037. PMID 18208346.
- ^ Jeon, Kwang W.; Berezney, Ronald (1995). Structural and functional organization of the nuclear matrix. Boston: Academic Press. pp. 214–7. ISBN 978-0-12-364565-4
- ^ “Solenoidal model for superstructure in chromatin”. Proceedings of the National Academy of Sciences of the United States of America 73 (6): 1897–901. (June 1976). Bibcode: 1976PNAS...73.1897F. doi:10.1073/pnas.73.6.1897. PMC 430414. PMID 1064861.
- ^ “Influence of histone H1 on chromatin structure”. Cell 12 (1): 101–7. (September 1977). doi:10.1016/0092-8674(77)90188-X. PMID 561660.
- ^ “Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin”. The Journal of Cell Biology 83 (2 Pt 1): 403–27. (November 1979). doi:10.1083/jcb.83.2.403. PMC 2111545. PMID 387806.
- ^ a b “What determines the folding of the chromatin fiber?”. Proceedings of the National Academy of Sciences of the United States of America 93 (20): 10548–55. (October 1996). Bibcode: 1996PNAS...9310548V. doi:10.1073/pnas.93.20.10548. PMC 38190. PMID 8855215.
- ^ “Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure”. Proceedings of the National Academy of Sciences of the United States of America 105 (26): 8872–7. (July 2008). Bibcode: 2008PNAS..105.8872R. doi:10.1073/pnas.0802336105. PMC 2440727. PMID 18583476.
- ^ “Heterogeneity of chromatin subunits in vitro and location of histone H1”. Nucleic Acids Research 3 (2): 477–92. (February 1976). doi:10.1093/nar/3.2.477. PMC 342917. PMID 1257057.
- ^ “Removal of histone H1 exposes a fifty base pair DNA segment between nucleosomes”. Biochemistry 15 (15): 3307–14. (July 1976). doi:10.1021/bi00660a022. PMID 952859.
- ^ a b “The structure of histone H1 and its location in chromatin”. Nature 288 (5792): 675–9. (December 1980). Bibcode: 1980Natur.288..675A. doi:10.1038/288675a0. PMID 7453800.
- ^ “Footprinting of linker histones H5 and H1 on the nucleosome”. The EMBO Journal 7 (12): 3685–91. (December 1988). doi:10.1002/j.1460-2075.1988.tb03250.x. PMC 454941. PMID 3208745.
- ^ “Nucleosomes, linker DNA, and linker histone form a unique structural motif that directs the higher-order folding and compaction of chromatin”. Proceedings of the National Academy of Sciences of the United States of America 95 (24): 14173–8. (November 1998). Bibcode: 1998PNAS...9514173B. doi:10.1073/pnas.95.24.14173. PMC 24346. PMID 9826673.
- ^ “The maternal histone H1 variant, H1M (B4 protein), is the predominant H1 histone in Xenopus pregastrula embryos”. Developmental Biology 161 (2): 425–39. (February 1994). doi:10.1006/dbio.1994.1042. PMID 8313993.
- ^ “Differential effect of H1 variant overexpression on cell cycle progression and gene expression”. Nucleic Acids Research 24 (3): 486–93. (February 1996). doi:10.1093/nar/24.3.486. PMC 145659. PMID 8602362.
- ^ “Effects of H1 histone variant overexpression on chromatin structure”. The Journal of Biological Chemistry 274 (53): 37950–6. (December 1999). doi:10.1074/jbc.274.53.37950. PMID 10608862.
- ^ “Histone H1 is essential for mitotic chromosome architecture and segregation in Xenopus laevis egg extracts”. The Journal of Cell Biology 169 (6): 859–69. (June 2005). doi:10.1083/jcb.200503031. PMC 2171634. PMID 15967810.
- ^ a b c “Functional comparison of H1 histones in Xenopus reveals isoform-specific regulation by Cdk1 and RanGTP”. Current Biology 20 (11): 1048–52. (June 2010). doi:10.1016/j.cub.2010.04.025. PMC 2902237. PMID 20471264.
- ^ “Linker histones are not essential and affect chromatin condensation in vivo”. Cell 82 (1): 47–56. (July 1995). doi:10.1016/0092-8674(95)90051-9. PMID 7606784.
- ^ “A single histone H1 isoform (H1.1) is essential for chromatin silencing and germline development in Caenorhabditis elegans”. Development 128 (7): 1069–80. (April 2001). PMID 11245572.
- ^ “Linker histone H1 is essential for Drosophila development, the establishment of pericentric heterochromatin, and a normal polytene chromosome structure”. Genes & Development 23 (4): 452–65. (February 2009). doi:10.1101/gad.1749309. PMC 2648648. PMID 19196654.
- ^ “Dynamic binding of histone H1 to chromatin in living cells”. Nature 408 (6814): 877–81. (December 2000). Bibcode: 2000Natur.408..877M. doi:10.1038/35048610. PMID 11130729.
- ^ “Condensed mitotic chromatin is accessible to transcription factors and chromatin structural proteins”. The Journal of Cell Biology 168 (1): 41–54. (January 2005). doi:10.1083/jcb.200407182. PMC 2171683. PMID 15623580.
- ^ “The dynamics of histone H1 function in chromatin”. Molecular Cell 17 (5): 617–20. (March 2005). doi:10.1016/j.molcel.2005.02.019. PMID 15749012.
- ^ “Histone H1 compacts DNA under force and during chromatin assembly”. Molecular Biology of the Cell 23 (24): 4864–71. (December 2012). doi:10.1091/mbc.E12-07-0518. PMC 3521692. PMID 23097493.
- ^ Cimini, Daniela, ed (September 2010). “Xenopus egg extracts increase dynamics of histone H1 on sperm chromatin”. PLOS One 5 (9): e13111. Bibcode: 2010PLoSO...513111F. doi:10.1371/journal.pone.0013111. PMC 2947519. PMID 20927327.
- ^ “Somatic linker histones cause loss of mesodermal competence in Xenopus”. Nature 389 (6649): 395–9. (September 1997). Bibcode: 1997Natur.389..395S. doi:10.1038/38755. PMID 9311783.
- ^ “Histone H1 variants differentially inhibit DNA replication through an affinity for chromatin mediated by their carboxyl-terminal domains”. Gene 292 (1–2): 173–81. (June 2002). doi:10.1016/S0378-1119(02)00675-3. PMID 12119111.
- ^ “Histone H1 diversity: bridging regulatory signals to linker histone function”. Gene 271 (1): 1–12. (June 2001). doi:10.1016/S0378-1119(01)00495-4. PMID 11410360.
- ^ “Cracking the enigmatic linker histone code”. Journal of Biochemistry 143 (3): 287–93. (March 2008). doi:10.1093/jb/mvn013. PMID 18234717.
- ^ “Histone H1 and its isoforms: contribution to chromatin structure and function”. Gene 431 (1–2): 1–12. (February 2009). doi:10.1016/j.gene.2008.11.003. PMID 19059319.
- ^ “H1 histones: current perspectives and challenges”. Nucleic Acids Research 41 (21): 9593–609. (November 2013). doi:10.1093/nar/gkt700. PMC 3834806. PMID 23945933.