核酸の三次構造
 ウィキペディアから無料の百科事典
ウィキペディアから無料の百科事典

核酸の三次構造(かくさんのさんじこうぞう)とは、核酸ポリマーの三次元的形状を指す[1]。RNAとDNAの分子は、分子認識や触媒などさまざまな機能を有する。このような機能を発揮するには正確な三次構造を取る必要がある。その構造は多様で一見複雑であるものの、簡単に認識できる三次構造モチーフがビルディングブロックとなって構成されている。ここではRNAとDNAの三次構造の最も一般的なモチーフの一部について記述するが、これらの情報は限られた数の既知構造に基づいている。新たなRNAやDNA分子の構造が解明されれば、さらに多くの三次構造モチーフが明らかとなるであろう。
らせん構造[編集]

二重らせん[編集]
二重らせんは生体のDNAで最も多くみられる三次構造であり、RNAもこの構造をとることができる。自然界で見つかるDNAのコンフォメーションはA-DNA、B-DNA、Z-DNAであると考えられている。ジェームズ・ワトソンとフランシス・クリックによって記載されたB型のDNAが、細胞内で最も多くみられる構造であると考えられている[2]。ワトソンとクリックは、この構造を半径 10 Å、ピッチ 34 Å、10塩基対ごとに1回転する二重らせんとして記載した[3]。溶液中では、二重らせんは10.4–10.5塩基対ごとに1回転する。このねじれの頻度は、各塩基が隣接塩基に及ぼすスタッキング相互作用に大きく依存している。RNAの二重らせんは、A型の構造に類似したコンフォメーションをとる。
A型、B型、Z型以外のコンフォメーションも可能である。事実、多くのコンフォメーションが発見されており、将来的に新たなDNA構造が見つかったときに使えるアルファベットはF、Q、U、V、Yだけしか残されていない[4][5]。しかし、これらの構造形式の大部分は人工的に作り出されたものであり、自然発生した生物学的な系では観察されていない。
三重鎖[編集]
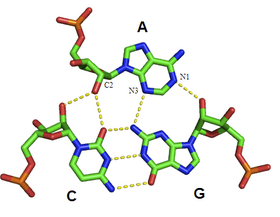
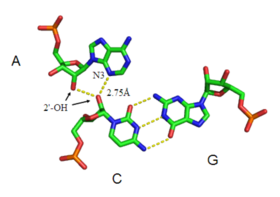
minor groove triplexは普遍的にみられるRNAの構造モチーフである。副溝(minor groove)との相互作用はリボースの2'-OH基を介して行われることが多いため、このRNAモチーフはDNAの三重鎖とは見た目が大きく異なる。minor groove triplexの例として最もよく見られるのはAマイナーモチーフ(A-minor motif)であり、アデニン塩基が副溝へ挿入される。しかし、このモチーフはアデニンに限定されているわけではなく、他の核酸塩基もRNAの副溝と相互作用することが観察されている。
副溝は挿入された塩基とほぼ完全な相補性を示す。これによって最適なファンデルワールス相互作用、広範囲の水素結合と疎水表面の埋没が可能となり、エネルギー的に極めて有利な相互作用が作り出される[8]。minor groove triplexはループとヘリックスを安定にパッキングすることができるため、グループIイントロン[9]、グループIIイントロン[10]、リボソームなどの巨大なリボヌクレオチド構造の主要な要素となっている。
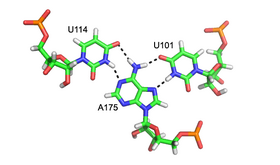
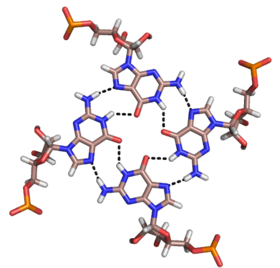
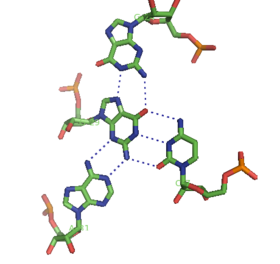
標準的なA型RNAの主溝(major groove)はかなり狭く、そのため副溝よりも三重鎖相互作用を形成しにくいが、いくつかのRNA構造ではmajor groove triplexが観察されている。これらの構造は塩基対とフーグスティーン型相互作用の組み合わせで構成されている。例えば、50SリボソームでみられるGGC triplex(GGC amino(N-2)-N-7, imino-carbonyl, carbonyl-amino(N-4); Watson-Crick)は、ワトソンクリック型のG-C塩基対に対し、挿入されたGが両塩基と擬フーグスティーン型の水素結合ネットワークを形成することで構成されている[11]。major groove triplexの他の注目すべき例としては、左の図で示されたグループIIイントロンの触媒コア[6]やヒトのテロメラーゼRNAで観察される触媒作用に必須な三重鎖[7]、そしてSAM-IIリボスイッチ[13]などがある。
DNA三重鎖も、B型DNAの主溝でフーグスティーン型または逆フーグスティーン型の水素結合を形成することによって可能である。
四重鎖[編集]
二重らせん、上述した三重鎖の他に、RNAとDNAはどちらも四重らせんを形成することができる。RNAの四重鎖には多様な構造が存在する。グアニン残基はフーグスティーン型水素結合によって“Hoogsteen ring”を形成し、四重鎖を形成する(グアニン四重鎖)[11]。G-CとA-U塩基対はワトソン・クリック型塩基対と副溝での非標準的塩基対との組み合わせによって四重鎖を形成することができる[14]。
マラカイトグリーンのアプタマーのコアには、さまざまな水素結合パターンからなる、一種の四重鎖構造が存在する[12]。四重鎖は何度か連続した繰り返し構造を形成することができ、非常に安定な構造が作り出される。
四重鎖領域の独特な構造は、生体システムでさまざまな機能を果たしている可能性がある。2つの重要な機能は、リガンドやタンパク質が結合する可能性と、DNAやRNAの全体的な三次構造を安定させる能力である。その強固な構造は、染色体のテロメア領域やmRNAのUTRで見られるように、転写や複製を阻害したり調節したりすることができる[15]。リガンドの結合には塩基の同一性が重要である。Gカルテットは通常カリウムイオンのような1価イオンを結合するが、他の塩基組成では他のリガンドを結合することができ、例えばU-U-C-U四重鎖はヒポキサンチンを結合する[14]。
これらの機能に加えて、細菌ではmRNA上のリボソーム結合部位周辺のグアニン四重鎖は遺伝子発現の調節因子として機能することがある[16]。まだin vivoでは発見されていない興味深い構造や機能が存在する可能性がある。
同軸的スタッキング[編集]

同軸的スタッキング(coaxial stacking)はヘリカル(らせん形)スタッキング(helical stacking)としても知られ、RNAの三次構造で高次構造を決定する主要な因子である。同軸的スタッキングは2つのRNA二重らせんが連続的なヘリックスを形成し、それら2つのヘリックスの相互作用面が塩基のスタッキングによって安定化されているときに生じる。同軸的スタッキングはtRNAPheの結晶構造の報告で記載された[18]。より最近では、同軸的スタッキングは、自己スプライシングを行うグループI、グループIIイントロンを含む、多くのリボザイムの高次構造で観察されている。同軸的スタッキングでよくみられるモチーフには、キッシングループ(kissing loop)相互作用とシュードノットがある。これらの相互作用の安定性は、“Turner’s rules” を適用することで予測することができる[19]。
1994年、WalterとTurnerは、ステムループ末端の4ヌクレオチドの突出部と短いオリゴマーの間でヘリックス-ヘリックス相互作用面を作り出すモデルシステムを用いて、ヘリックス-ヘリックス相互作用面内の最近接スタッキング相互作用の自由エネルギーへの寄与の程度を決定した。彼らの実験は、2つのヘリックス間の塩基スタッキングの熱力学的な寄与は、標準的な二重らせん形成時の熱力学的寄与(形成されるヘリックスの熱力学的安定性を予測する最近接相互作用)にきわめて近似的なものであることを確証した。最近接相互作用の相対的安定性は、エネルギー的に有利な同軸的スタッキングを既知二次構造に基づいて予想する際に利用することができる。WalterとTurnerは、同軸的スタッキングによる寄与を考慮に入れることで、RNA構造予測の正確さが平均して67%から74%へ改善することを見出した[20]。
最もよく研究されているRNA三次構造には、同軸的スタッキングの例が含まれている。よく知られた例としては、tRNAPhe、グループIイントロン、グループIIイントロン、そしてリボソームRNAがある。tRNAの結晶構造解析によって、アミノ酸アクセプターステムとTアームのスタッキング、Dアームとアンチコドンステムのスタッキングという、同軸的スタッキングによって伸長したヘリックスが存在することが明らかにされた。これらのtRNA内部の相互作用によって、アンチコドンステムとアクセプターステムは直交した向きとなり、機能的なL字型の三次構造が形成される[18]。グループIイントロンでは、P4、P6ヘリックスが同軸的スタッキングを行うことが、生化学的[21]、結晶学的手法の組み合わせによって示されている。P4-P6ヘリックスの結晶構造では、同軸的スタッキングがどのようにRNAヘリックスを三次構造へとパッキングし安定化するかが詳細に示された[22]。Oceanobacillus iheyensisの自己スプライシンググループIIイントロンでは、IA、IBステムが同軸的スタッキングを行い、five-way junctionの構成要素となるヘリックスの相対的配向に寄与している[6]。この配向は、機能的リボザイムの活性部位の適切なフォールディングを促進する。リボソームには同軸的スタッキングの多数の例が含まれ、その中には70 bpもの長さのものも含まれる[23]。

同軸的スタッキングでよくみられる2つのモチーフは、キッシングループとシュードノットである。キッシングループ相互作用では、2つのヘアピンの一本鎖のループ領域が塩基対形成によって相互作用し、同軸的スタッキングによってヘリックスが合成される。この構造ではそれぞれのループの全てのヌクレオチドが塩基対形成とスタッキング相互作用に参加することができる。このモチーフは、LeeとCrothersによってNMR解析による可視化と研究が行われた[24]。シュードノットモチーフは、ヘアピンループの一本鎖領域が同じRNA鎖の上流または下流の配列と塩基対を形成することで生じる。その結果生じた2つの二重らせんが互いにスタッキングし、安定な同軸的スタッキングヘリックスが合成される。シュードノットの例としては極めて安定性が高いD型肝炎ウイルスリボザイムがあり、その主鎖は全体で二重のシュードノット型のトポロジーとなっている[25]。
合理的設計がなされたDNA構造では、同軸的スタッキングに似た効果がみられる。DNAオリガミ構造は、平滑末端が露出した二重らせんを多数含んでいる。これらの構造では、露出した平滑末端が疎水的なスタッキング相互作用のために互いに接着することが観察されている[26]。
他のモチーフ[編集]
テトラループ-受容体相互作用[編集]

テトラループとその受容体間の相互作用は、テトラループモチーフのループ部分のヌクレオチドとRNA二重らせん内部の受容体モチーフにおける、塩基対形成とスタッキング相互作用の組み合わせであり、三次元的な相互作用を作り出すことでRNA分子全体の三次元的フォールドを安定化している。また、テトラループはDNA二重らせん中でも可能な構造である[28]。
ステムループのサイズや配列はさまざまであるが、テトラループの4つのヌクレオチドは非常に共通している。それらは配列に基づいて通常3つのカテゴリーのいずれかに属し、CUYG、UNCG、GNRAテトラループという3つのファミリーが存在する[29]。これらのテトラループでは2番目と3番目のヌクレオチドがRNA鎖のターンを形成し、1番目と4番目のヌクレオチドが塩基対を形成してステムループ構造を安定化する。一般的にテトラループの安定性は、ループ内部の塩基組成とこの"closing base pair"の構成に依存することが決定されている[30]。GNRAファミリーのテトラループは、テトラループ-受容体相互作用で最もよく観察されるタイプである。

「テトラループ受容体モチーフ」は、テトラループ内の塩基がRNAの二次構造上で離れた位置にあるステムループ配列との水素結合する、長距離の三次元的相互作用である[31][32]。水素結合に加えて、スタッキング相互作用はこれらの三次元的相互作用の重要な要素となる。例えば、GNRAテトラループの相互作用では、テトラループの2番目のヌクレオチドは受容体内部のA-プラットフォームモチーフへ直接スタッキングする[22]。テトラループとその受容体の配列は、さまざまテトラループのアイソフォームとその受容体との間で同じタイプの三次元的接触が起こるよう、しばしば共変化が起こっている[33]。
自己スプライシンググループIイントロンの構造や機能はテトラループ受容体モチーフに依存している[22][32]。特に、典型的なGAAAモチーフの3つのアデニン塩基は受容体ヘリックスの上にスタッキングし、受容体と複数の水素結合を形成して安定化する。GAAA配列の最初のアデニンは、受容体のAU塩基と三重鎖構造を形成する。2番目のアデニンは、同じウリジンとの水素結合と、受容体の2'-OH基、GAAAテトラループのグアニンとの相互作用によって安定化される。3番目のアデニンは三重鎖構造を形成する。
A-マイナーモチーフ[編集]
A-マイナーモチーフは普遍的にみられるRNAの三次構造モチーフであり、対合していないヌクレオシドがRNA二重らせんの副溝に挿入されることで形成される。それゆえ、minor groove triplexの一例である。グアノシン、シトシン、ウリジンも副溝での三重鎖相互作用を形成することはできるものの、アデニンによる相互作用が極めて一般的である。アデニンの場合、挿入塩基のN1-C2-N3端は、二重らせん塩基の他に、一方または双方の2’-OH基とも水素結合を形成する。挿入塩基を受け入れるのは、多くの場合G-C塩基対である。
A-マイナーモチーフは、ワトソン-クリック型塩基対の2'-OHに対する挿入塩基の相対的な位置に基づいて、0型からIII型の4つのクラスに分類される[8]。I・II型のA-マイナーモチーフでは、アデニンのN3は二重らせんの副溝内部へ深く挿入され、塩基対とは良い形状相補性を示す。0・III型と異なり、I・II型相互作用はその水素結合パターンからアデニン特異的である。III型相互作用では、挿入塩基のO2'とN3の双方が二重らせんの副溝とより離れた位置で結合する。0・III型モチーフの相互作用は1つの2’-OH基によって媒介され、より弱く非特異的である。
A-マイナーモチーフはリボソームで最もよく見られるRNAの構造モチーフであり、tRNAの23S rRNAへの結合にも寄与している[35]。グループIIイントロンのコアでみられるように、ほとんどの場合ループやヘリックス部分で二重らせん相互作用を安定化している[6]。
A-マイナーモチーフの興味深い例は、アンチコドンの認識における役割である。リボソームはコドン-アンチコドンの正確な対合と不正確な対合を見分けなければならず、その過程の一部は副溝へのアデニン塩基の挿入によって行われている。不正確なコドン-アンチコドン対合ではヘリックスの形状が歪み、A-マイナー相互作用による結合安定化が妨げられ、不正確なtRNAの解離速度が増加する[36]。
23S rRNA中のA-マイナーモチーフの分析によって、構造に依存した階層的ネットワークが存在することが明らかとなり、リボソームの進化や現代の細菌の大サブユニットの発展をもたらしたイベントの順序との関連が示唆されている[37]。
リボースジッパー[編集]

リボースジッパーは、2つのRNA鎖がリボースの2’-OH基の水素結合によって保持される三次構造エレメントである。2’-OH基は水素結合の供与体としても受容体としても機能し、他方の鎖の2’-OH基と二股の水素結合を形成する[38][39]。
リボースジッパーは多数の形状が報告されているが、一般的なタイプは2つの近接した糖の2'-OH基の間で4つの水素結合が形成されるものである。一般的にリボースジッパーは、別々のRNA鎖間の相互作用を安定させる配置で形成される[40]。リボースジッパーはしばしば極めて配列特異性の低いステムループ相互作用として観察される。しかしリボソームの大サブユニットと小サブユニットでは、リボースジッパーはCC/AA配列となる(一方の鎖の2つのシトシンが他方の鎖の2つのアデニンと対合する)傾向がある。
金属イオンの役割[編集]
機能的なRNAは多くの場合、ふらふらとした直鎖ではなく、フォールディングした安定な三次元的形状を有する分子である[42]。カチオンは、RNAの三次構造の熱力学的な安定化に必須である。RNAに結合する金属カチオンは、1価、2価、そして3価の場合もある。カリウム(K+)は、RNAに結合する最も一般的な1価イオンである。RNAに結合する一般的な2価イオンは、マグネシウム(Mg2+)である。ナトリウム(Na+)、カルシウム(Ca2+)、マンガン(Mn2+)など他のイオンもin vivoとin vitroでRNAに結合することが判明している。スペルミジンやスペルミンといった多価有機カチオンも細胞内に存在し、RNAのフォールディングに重要な寄与をしている。コバルトヘキサミンや、テルビウム(Tb3+)などのランタノイドイオンのような3価イオンは、RNAへの金属の結合を研究する有用な実験的ツールである[43][44]。
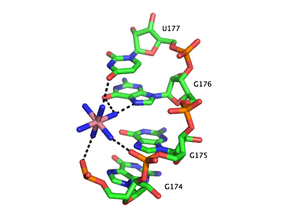
金属イオンは複数の方法でRNAと相互作用する。あるイオンはRNAの主鎖と散在的な結合を行い、静電的相互作用を遮蔽する。この電荷遮蔽は多くの場合1価イオンによって行われる。一方、特定の部位に結合するイオンは、RNAの三次構造の特定のエレメントの安定化を行う。その相互作用は、金属結合を水分子が媒介するかどうかによって2つのカテゴリーにさらに分類される。外圏型(outer sphere)の相互作用は、金属イオンを囲む水分子によって媒介される。例えば、マグネシウム六水和物は特定のRNA三次構造モチーフの主鎖のグアノシンを介して相互作用し安定化する。逆に、内圏型(inner sphere)の相互作用は、直接金属イオンによって媒介される。RNAは多くの場合多段階のフォールディングを行い、その各段階は異なるタイプのカチオンによって安定化される。後期の段階はRNAの三次構造の形成を伴い、主にマグネシウムなどの2価イオンの結合によって安定化されるが、カリウムイオンの結合も寄与している可能性がある。
金属結合部位は多くの場合RNA二重らせんの深く狭い主溝に局在し、プリン塩基のフーグスティーンエッジ(フーグスティーン型塩基対を形成する側)に配位する。特に、金属カチオンは主鎖がねじれ、リン酸が密にパッキングし濃密な負電荷となる部位を安定化する。RNA二重らせん中の金属イオン結合モチーフがいくつか結晶構造中に同定されている。例えば、Tetrahymena thermophilaのグループIイントロンのP4-P6ドメイン中にはタンデムなG-Uゆらぎ塩基対とG-Aミスマッチからなるイオン結合部位がいくつか存在し、そこではグアノシンのフーグスティーンエッジのO6とN7を介して2価カチオンが相互作用する[45][46][47]。テトラヒメナのグループIイントロン中の他のイオン結合モチーフとしてはA-Aプラットフォームモチーフがあり、RNAの同じ鎖中で連続したアデノシンが非典型的な擬似塩基対を形成する[48]。タンデムG-Uモチーフとは異なり、A-Aプラットフォームモチーフは1価カチオンを好んで結合する。これらのモチーフの多くでは、1価または2価カチオンが存在しないときには構造の柔軟性が大きく増加するか、その三次構造を失う。
2価金属イオン、特にマグネシウムは、遺伝的組換えにおけるホリデイジャンクション中間体などのDNAジャンクション構造に重要であることが判明している。マグネシウムイオンはジャンクション中の負に帯電したリン酸基を遮蔽し、近接して配置されてスタッキングしたコンフォメーションを取ることを可能にする[49]。マグネシウムは、double crossover motifなどDNAナノテクノロジーで利用される人工的にデザインされた構造中でのジャンクションの安定化にも重要である[50]。
出典[編集]
- ^ IUPAC, Compendium of Chemical Terminology, 2nd ed. (the "Gold Book") (1997). オンライン版: (2006-) "tertiary structure".
- ^ “The structure of DNA in the nucleosome core”. Nature 423 (6936): 145–50. (May 2003). Bibcode: 2003Natur.423..145R. doi:10.1038/nature01595. PMID 12736678.
- ^ “Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid” (PDF). Nature 171 (4356): 737–8. (April 1953). Bibcode: 1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692.
- ^ Bansal M (2003). “DNA structure: Revisiting the Watson-Crick double helix”. Current Science 85 (11): 1556–1563.
- ^ “A glossary of DNA structures from A to Z”. Acta Crystallogr D 59 (4): 620–626. (2003). doi:10.1107/S0907444903003251. PMID 12657780.
- ^ a b c d PDB: 3BWP; “Crystal structure of a self-spliced group II intron”. Science 320 (5872): 77–82. (April 2008). Bibcode: 2008Sci...320...77T. doi:10.1126/science.1153803. PMC 4406475. PMID 18388288.; rendered with PyMOL
- ^ a b PDB: 2K95; “Solution structure and dynamics of the wild-type pseudoknot of human telomerase RNA”. J. Mol. Biol. 384 (5): 1249–61. (December 2008). doi:10.1016/j.jmb.2008.10.005. PMC 2660571. PMID 18950640.; rendered with PyMOL
- ^ a b “A universal mode of helix packing in RNA”. Nat. Struct. Biol. 8 (4): 339–43. (April 2001). doi:10.1038/86221. PMID 11276255.
- ^ “A minor groove RNA triple helix within the catalytic core of a group I intron”. Nat. Struct. Biol. 5 (12): 1037–42. (December 1998). doi:10.1038/4146. PMID 9846872.
- ^ “A tertiary interaction that links active-site domains to the 5' splice site of a group II intron”. Nature 406 (6793): 315–8. (July 2000). doi:10.1038/35018589. PMID 10917534.
- ^ a b c PDB: 1RAU; “Solution structure of an unusually stable RNA tetraplex containing G- and U-quartet structures”. Biochemistry 31 (36): 8406–14. (September 1992). doi:10.1021/bi00151a003. PMID 1382577.; rendered with PyMOL
- ^ a b PDB: 1FIT; “2.8 A crystal structure of the malachite green aptamer”. J. Mol. Biol. 301 (1): 117–28. (August 2000). doi:10.1006/jmbi.2000.3951. PMID 10926496.; rendered with PyMOL
- ^ “Structure of the SAM-II riboswitch bound to S-adenosylmethionine”. Nat. Struct. Mol. Biol. 15 (2): 177–82. (February 2008). doi:10.1038/nsmb.1371. PMID 18204466.
- ^ a b “Structure of a natural guanine-responsive riboswitch complexed with the metabolite hypoxanthine”. Nature 432 (7015): 411–5. (November 2004). Bibcode: 2004Natur.432..411B. doi:10.1038/nature03037. PMID 15549109.
- ^ “Functional and dysfunctional roles of quadruplex DNA in cells”. Chem. Biol. 8 (3): 221–30. (March 2001). doi:10.1016/S1074-5521(01)00007-2. PMID 11306347.
- ^ “Preferential binding of fd gene 5 protein to tetraplex nucleic acid structures”. J. Mol. Biol. 301 (3): 575–84. (August 2000). doi:10.1006/jmbi.2000.3991. PMID 10966771.
- ^ PDB: 6tna; “Crystal structure of yeast phenylalanine transfer RNA. I. Crystallographic refinement”. J. Mol. Biol. 123 (4): 607–30. (August 1978). doi:10.1016/0022-2836(78)90209-7. PMID 357742.; rendered via PyMOL.
- ^ a b “Structural domains of transfer RNA molecules”. Science 194 (4267): 796–806. (November 1976). Bibcode: 1976Sci...194..796Q. doi:10.1126/science.790568. PMID 790568.
- ^ Turner, Douglas H.; Mathews, David H. (2010-1). “NNDB: the nearest neighbor parameter database for predicting stability of nucleic acid secondary structure”. Nucleic Acids Research 38 (Database issue): D280–D282. doi:10.1093/nar/gkp892. ISSN 0305-1048. PMC 2808915. PMID 19880381.
- ^ “Coaxial stacking of helixes enhances binding of oligoribonucleotides and improves predictions of RNA folding”. Proc. Natl. Acad. Sci. U.S.A. 91 (20): 9218–22. (September 1994). Bibcode: 1994PNAS...91.9218W. doi:10.1073/pnas.91.20.9218. PMC 44783. PMID 7524072.
- ^ “Coaxially stacked RNA helices in the catalytic center of the Tetrahymena ribozyme”. Science 265 (5179): 1709–12. (September 1994). Bibcode: 1994Sci...265.1709M. doi:10.1126/science.8085157. PMID 8085157.
- ^ a b c “Crystal structure of a group I ribozyme domain: principles of RNA packing”. Science 273 (5282): 1678–85. (September 1996). Bibcode: 1996Sci...273.1678C. doi:10.1126/science.273.5282.1678. PMID 8781224.
- ^ Noller HF (September 2005). “RNA structure: reading the ribosome”. Science 309 (5740): 1508–14. Bibcode: 2005Sci...309.1508N. doi:10.1126/science.1111771. PMID 16141058.
- ^ “The solution structure of an RNA loop-loop complex: the ColE1 inverted loop sequence”. Structure 6 (8): 993–1005. (August 1998). doi:10.1016/S0969-2126(98)00101-4. PMID 9739090.
- ^ “Crystal structure of a hepatitis delta virus ribozyme”. Nature 395 (6702): 567–74. (October 1998). Bibcode: 1998Natur.395..567F. doi:10.1038/26912. PMID 9783582.
- ^ Rothemund, Paul W. K. (2006). “Folding DNA to create nanoscale shapes and patterns”. Nature 440 (7082): 297–302. Bibcode: 2006Natur.440..297R. doi:10.1038/nature04586. ISSN 0028-0836. PMID 16541064.
- ^ a b c d PDB: 1GID; “Crystal structure of a group I ribozyme domain: principles of RNA packing”. Science 273 (5282): 1678–85. (September 1996). Bibcode: 1996Sci...273.1678C. doi:10.1126/science.273.5282.1678. PMID 8781224.; rendered with PyMOL
- ^ “Selection for thermodynamically stable DNA tetraloops using temperature gradient gel electrophoresis reveals four motifs: d(cGNNAg), d(cGNABg),d(cCNNGg), and d(gCNNGc)”. Biochemistry 41 (48): 14281–92. (December 2002). doi:10.1021/bi026479k. PMID 12450393.
- ^ “Remarkable morphological variability of a common RNA folding motif: the GNRA tetraloop-receptor interaction”. J. Mol. Biol. 266 (3): 493–506. (February 1997). doi:10.1006/jmbi.1996.0810. PMID 9067606.
- ^ “Evidence that folding of an RNA tetraloop hairpin is less cooperative than its DNA counterpart”. Biochemistry 43 (25): 7992–8. (June 2004). doi:10.1021/bi049350e. PMID 15209494.
- ^ Nucleic Acids in Chemistry and Biology. Cambridge, UK: RSC Pub. (2006). ISBN 0-85404-654-2
- ^ a b “Involvement of a GNRA tetraloop in long-range RNA tertiary interactions”. J. Mol. Biol. 236 (5): 1271–6. (March 1994). doi:10.1016/0022-2836(94)90055-8. PMID 7510342.
- ^ “Modelling of the three-dimensional architecture of group I catalytic introns based on comparative sequence analysis”. J. Mol. Biol. 216 (3): 585–610. (December 1990). doi:10.1016/0022-2836(90)90386-Z. PMID 2258934.
- ^ a b PDB: 1FFK; “The complete atomic structure of the large ribosomal subunit at 2.4 A resolution”. Science 289 (5481): 905–20. (August 2000). Bibcode: 2000Sci...289..905B. doi:10.1126/science.289.5481.905. PMID 10937989.; rendered with PyMOL
- ^ “RNA tertiary interactions in the large ribosomal subunit: the A-minor motif”. Proc. Natl. Acad. Sci. U.S.A. 98 (9): 4899–903. (April 2001). Bibcode: 2001PNAS...98.4899N. doi:10.1073/pnas.081082398. PMC 33135. PMID 11296253.
- ^ “Recognition of the codon-anticodon helix by ribosomal RNA”. Science 285 (5434): 1722–5. (September 1999). doi:10.1126/science.285.5434.1722. PMID 10481006.
- ^ “A hierarchical model for evolution of 23S ribosomal RNA”. Nature 457 (7232): 977–80. (February 2009). Bibcode: 2009Natur.457..977B. doi:10.1038/nature07749. PMID 19225518.
- ^ “Tertiary Motifs in RNA Structure and Folding”. Angew. Chem. Int. Ed. Engl. 38 (16): 2326–2343. (August 1999). doi:10.1002/(SICI)1521-3773(19990816)38:16<2326::AID-ANIE2326>3.0.CO;2-3. PMID 10458781.
- ^ “Sequence and structural conservation in RNA ribose zippers”. J. Mol. Biol. 320 (3): 455–74. (July 2002). doi:10.1016/S0022-2836(02)00515-6. PMID 12096903.
- ^ PDB: 3IGI; “Tertiary architecture of the Oceanobacillus iheyensis group II intron”. RNA 16 (1): 57–69. (January 2010). doi:10.1261/rna.1844010. PMC 2802037. PMID 19952115.; rendered using PyMOL.
- ^ PDB: 1ZZN; “Structural evidence for a two-metal-ion mechanism of group I intron splicing”. Science 309 (5740): 1587–90. (September 2005). Bibcode: 2005Sci...309.1587S. doi:10.1126/science.1114994. PMID 16141079.; rendered with PyMOL
- ^ “Visualizing the higher order folding of a catalytic RNA molecule”. Science 251 (4992): 401–7. (January 1991). Bibcode: 1991Sci...251..401C. doi:10.1126/science.1989074. PMID 1989074.
- ^ Pyle AM (September 2002). “Metal ions in the structure and function of RNA”. J. Biol. Inorg. Chem. 7 (7–8): 679–90. doi:10.1007/s00775-002-0387-6. PMID 12203005.
- ^ Morrow, Janet R.; Andolina, Christopher M. (2012). “Chapter 6. Spectroscopic Investigations of Lanthanide Ion Binding to Nucleic Acids”. Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. 10. Springer. pp. 171–197. doi:10.1007/978-94-007-2172-2_6
- ^ “Metal-binding sites in the major groove of a large ribozyme domain”. Structure 4 (10): 1221–9. (October 1996). doi:10.1016/S0969-2126(96)00129-3. PMID 8939748.
- ^ “Solution structure of a metal-binding site in the major groove of RNA complexed with cobalt (III) hexammine”. Structure 5 (5): 713–21. (May 1997). doi:10.1016/S0969-2126(97)00225-6. PMID 9195889.
- ^ “Solution structure of Cobalt(III)hexammine complexed to the GAAA tetraloop, and metal-ion binding to G·A mismatches”. J. Mol. Biol. 295 (5): 1211–23. (February 2000). doi:10.1006/jmbi.1999.3421. PMID 10653698.
- ^ “Modelling ion binding to AA platform motifs in RNA: a continuum solvent study including conformational adaptation”. Nucleic Acids Res. 29 (19): 3910–8. (October 2001). doi:10.1093/nar/29.19.3910. PMC 60250. PMID 11574672.
- ^ “A pivotal role for the structure of the Holliday junction in DNA branch migration”. The EMBO Journal 14 (8): 1819–26. (April 1995). doi:10.1002/j.1460-2075.1995.tb07170.x. PMC 398275. PMID 7737132.
- ^ “DNA double-crossover molecules”. Biochemistry 32 (13): 3211–20. (April 1993). doi:10.1021/bi00064a003. PMID 8461289.