増殖細胞核抗原
 ウィキペディアから無料の百科事典
ウィキペディアから無料の百科事典

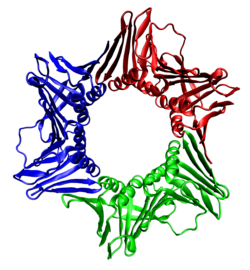
増殖細胞核抗原(ぞうしょくさいぼうかくこうげん、英: proliferating cell nuclear antigen、略称: PCNA)は、真核生物細胞においてDNAポリメラーゼδのプロセシビティ因子として作用するDNAクランプであり、DNA複製に必要不可欠である。PCNAはホモ三量体を形成し、DNAを取り囲むことでプロセシビティを高め、DNA複製、DNA修復、クロマチンリモデリング、エピジェネティクスに関与するタンパク質をリクルートするための足場として機能する[5]。
多くのタンパク質は、PIP(PCNA-interacting peptide)ボックス[6]とAPIM(AlkB homologue 2 PCNA interacting motif)[7]という2つのPCNA相互作用モチーフを介してPCNAと相互作用する。PIPボックスを介してPCNAに結合するタンパク質が主にDNA複製に関与しているのに対し、APIMを介してPCNAに結合するタンパク質は主に遺伝毒性ストレスとの関係で重要である[8]。
機能[編集]
PCNA遺伝子にコードされるPCNAタンパク質は核内に存在し、DNAポリメラーゼδ(Pol δ)のコファクターである。PCNAはホモ三量体の形で機能し、DNA複製時にリーディング鎖合成のプロセシビティの向上を補助する。また、DNA損傷に応答してPCNAはユビキチン化され、RAD6依存的DNA修復経路に関与する。この遺伝子には同一のタンパク質をコードする2種類の転写産物が見つかっている。この遺伝子の偽遺伝子が4番染色体とX染色体に存在する[9]。
DNA合成時における核内での発現[編集]
PCNAはもともと、細胞周期のDNA合成期(S期)に細胞核で発現している抗原として同定された[10]。タンパク質の配列の一部が解析され、それをもとにcDNAクローンが単離された[11]。PCNAはPol δのDNAへの保持を補助する。PCNAは複製因子C(RFC)の作用によってDNAへのクランプを形成する[12][13]。RFCはAAA+ファミリーのATPアーゼのヘテロ五量体型のメンバーである。PCNAの発現は、転写因子E2Fを含む複合体の制御下にある[14][15]。
DNA修復における役割[編集]
DNAポリメラーゼδやεはDNA修復時に除去された損傷DNA鎖の再合成に関与しているため、PCNAはDNA合成とDNA修復の双方に重要である[16][17]。
PCNAは複製後修復(PRR)と呼ばれるDNA損傷トレランス経路にも関与している[18]。PRRには2つのサブ経路が存在し、損傷乗り越え(translesion)経路は損傷したDNA塩基を活性部位に取り込むことができる特殊なDNAポリメラーゼによって行われ(通常の複製ポリメラーゼは停止してしまう)、鋳型乗り換え(template switch)経路では相同組換え装置のリクルートによって損傷部位の迂回が行われると考えられている。PCNAはこれらの経路の活性化や、どちらの経路が利用されるかの選択に重要である。PCNAはユビキチン化による翻訳後修飾が行われる[19]。PCNAのリジン164番のモノユビキチン化は損傷乗り越え経路を活性化する。このモノユビキチンに対して非典型的なリジン63番連結型のポリユビキチン化が行われると、鋳型乗り換え経路が活性化されると考えられている[19]。さらに、PCNAのリジン164番(と程度は低いもののリジン127番)のSUMO化は鋳型乗り換え経路を阻害する[19]。SUMO化されたPCNAはSrs2と呼ばれるDNAヘリカーゼをリクルートし[20]、相同組換えの開始に重要なRad51ヌクレオタンパク質フィラメントを破壊することでこの拮抗的な作用を示す。
相互作用[編集]
PCNAには、DNAポリメラーゼ、クランプローダー、フラップエンドヌクレアーゼ、DNAリガーゼ、トポイソメラーゼ、ライセンス化因子、E3ユビキチンリガーゼ、E2 SUMO結合酵素、ヘリカーゼ(ATPアーゼ)、ミスマッチ修復酵素、塩基除去修復酵素、ヌクレオチド除去修復酵素、ポリ(ADP-リボース)ポリメラーゼ、ヒストンシャペロン、クロマチンリモデリング因子、ヒストンアセチル化酵素、ヒストン脱アセチル化酵素、DNAメチルトランスフェラーゼ、姉妹染色分体接着因子、プロテインキナーゼ、細胞周期調節因子、アポトーシス関連因子など、多くのタンパク質が結合する[21]。
より具体的には、次の挙げる因子との相互作用が示されている。
- ALKBH2[7]
- ANXA2[22]
- CAF-1[23][24][25]
- CDC25C[26]
- CHTF18[22]
- CCND1[27][28]
- CCNO[22][29]
- CDK4[28][30]
- CDKN1C[31]
- DNMT1[32][33][34]
- EP300[35]
- ESCO2[36]
- FBH1[37]
- FEN1[38][39][40][41][42][43][44]
- GADD45A[45][46][47][48][49]
- GADD45G[50][51]
- GTF2I[7]
- HDAC1[52]
- HUS1[53]
- ING1[54]
- KCTD13[55]
- Ku70[22][56]
- Ku80[22][56][57]
- MCL1[58]
- MSH3[22][59][60]
- MSH6[22][59][60]
- MUTYH[61]
- P21[31][40][44][62][63][64][65][66]
- PCLAF[44]
- POLD2[67]
- POLD3[22][68]
- POLDIP2[69]
- POLH[70]
- POLL[71][72][73]
- RFC1[22][62][74][75][76]
- RFC2[22][77][78]
- RFC3[22][79]
- RFC4[22][77]
- RFC5[22][75][77]
- UBC[80][81][82]
- WRN[83][84]
- XPA[85]
- XRCC1[86]
- YBX1[87]
- ZRANB3[88]
利用[編集]
PCNAに対する抗体またはKi-67と呼ばれるモノクローナル抗体が、星細胞腫などさまざまな新生物のグレーディングに利用される。これらは診断や予後の判定に利用できる。抗体標識によるPCNAの核内分布のイメージングは、細胞周期のS期の初期、中期、後期を区別するために用いることができる[89]。しかしながら、抗体を用いる際の重要な制限として、細胞を固定する必要があるためにアーティファクトが生じる可能性がある。
一方、生細胞における複製と修復のダイナミクスの研究は、PCNAと蛍光タンパク質などとの融合タンパク質の導入によって行われる場合がある。また、トランスフェクションの必要性やトランスフェクションが困難であったり細胞の寿命が短いといった問題を回避するために、細胞透過性ペプチドによる複製・修復マーカーが利用される場合がある。これらのペプチドは、生きた組織においてin situで使用でき、複製中の細胞と修復中の細胞を区別することもできるという利点がある[90]。
PCNAは、がん治療における治療標的としての可能性がある[91]。
出典[編集]
- ^ a b c GRCh38: Ensembl release 89: ENSG00000132646 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000027342 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ “PCNA, the maestro of the replication fork.”. Cell 129 (4): 665–79. (May 18, 2007). doi:10.1016/j.cell.2007.05.003. PMID 17512402.
- ^ “PCNA binding through a conserved motif.”. BioEssays 20 (3): 195–9. (Mar 1998). doi:10.1002/(sici)1521-1878(199803)20:3<195::aid-bies2>3.0.co;2-r. PMID 9631646.
- ^ a b c “Identification of a novel, widespread, and functionally important PCNA-binding motif.”. The Journal of Cell Biology 186 (5): 645–54. (Sep 7, 2009). doi:10.1083/jcb.200903138. PMC 2742182. PMID 19736315.
- ^ “Regulation of PCNA-protein interactions for genome stability.”. Nature Reviews Molecular Cell Biology 14 (5): 269–82. (May 2013). doi:10.1038/nrm3562. PMID 23594953.
- ^ “Entrez Gene: PCNA proliferating cell nuclear antigen”. 2022年1月10日閲覧。
- ^ “PCNA and Ki67 expression in breast carcinoma: correlations with clinical and biological variables”. J. Clin. Pathol. 45 (5): 416–419. (1992). doi:10.1136/jcp.45.5.416. PMC 495304. PMID 1350788.
- ^ “Molecular cloning of cDNA coding for rat proliferating cell nuclear antigen (PCNA)/cyclin”. EMBO J. 6 (3): 637–42. (1987). doi:10.1002/j.1460-2075.1987.tb04802.x. PMC 553445. PMID 2884104.
- ^ “Structural analysis of a eukaryotic sliding DNA clamp-clamp loader complex”. Nature 429 (6993): 724–730. (2004). Bibcode: 2004Natur.429..724B. doi:10.1038/nature02585. PMID 15201901.
- ^ “Studies on the interactions between human replication factor C and human proliferating cell nuclear antigen”. Proc. Natl. Acad. Sci. U.S.A. 96 (5): 1869–1874. (1999). Bibcode: 1999PNAS...96.1869Z. doi:10.1073/pnas.96.5.1869. PMC 26703. PMID 10051561.
- ^ “Two E2F elements regulate the proliferating cell nuclear antigen promoter differently during leaf development”. Plant Cell 14 (12): 3225–3236. (2002). doi:10.1105/tpc.006403. PMC 151214. PMID 12468739.
- ^ “HER2 Signaling Drives DNA Anabolism and Proliferation through SRC-3 Phosphorylation and E2F1-Regulated Genes”. Cancer Res 76 (6): 1463–75. (15 March 2016). doi:10.1158/0008-5472.CAN-15-2383. PMC 4794399. PMID 26833126.
- ^ “Proliferating cell nuclear antigen is required for DNA excision repair”. Cell 69 (2): 367–74. (April 1992). doi:10.1016/0092-8674(92)90416-A. PMID 1348971.
- ^ “Nuclear dynamics of PCNA in DNA replication and repair”. Mol. Cell. Biol. 25 (21): 9350–9359. (2005). doi:10.1128/MCB.25.21.9350-9359.2005. PMC 1265825. PMID 16227586.
- ^ “Gaps and forks in DNA replication: Rediscovering old models”. DNA Repair (Amst.) 5 (12): 1495–1498. (December 2006). doi:10.1016/j.dnarep.2006.07.002. PMID 16956796.
- ^ a b c “RAD6-dependent DNA repair is linked to modification of PCNA by ubiquitin and SUMO”. Nature 419 (6903): 135–141. (September 2002). Bibcode: 2002Natur.419..135H. doi:10.1038/nature00991. PMID 12226657.
- ^ “SUMO-modified PCNA recruits Srs2 to prevent recombination during S phase”. Nature 436 (7049): 428–33. (July 2005). Bibcode: 2005Natur.436..428P. doi:10.1038/nature03665. PMID 15931174.
- ^ “PCNA, the maestro of the replication fork”. Cell 129 (4): 665–679. (2007). doi:10.1016/j.cell.2007.05.003. PMID 17512402.
- ^ a b c d e f g h i j k l m “A proteomics approach to identify proliferating cell nuclear antigen (PCNA)-binding proteins in human cell lysates. Identification of the human CHL12/RFCs2-5 complex as a novel PCNA-binding protein”. J. Biol. Chem. 277 (43): 40362–7. (October 2002). doi:10.1074/jbc.M206194200. PMID 12171929.
- ^ “A DNA binding winged helix domain in CAF-1 functions with PCNA to stabilize CAF-1 at replication forks”. Nucleic Acids Research 44 (11): 5083–94. (June 2016). doi:10.1093/nar/gkw106. PMC 4914081. PMID 26908650.
- ^ “A CAF-1-PCNA-mediated chromatin assembly pathway triggered by sensing DNA damage”. Molecular and Cellular Biology 20 (4): 1206–18. (February 2000). doi:10.1128/mcb.20.4.1206-1218.2000. PMC 85246. PMID 10648606.
- ^ “Two fundamentally distinct PCNA interaction peptides contribute to chromatin assembly factor 1 function”. Molecular and Cellular Biology 29 (24): 6353–65. (December 2009). doi:10.1128/MCB.01051-09. PMC 2786881. PMID 19822659.
- ^ “Cdc25C interacts with PCNA at G2/M transition”. Oncogene 21 (11): 1717–26. (March 2002). doi:10.1038/sj.onc.1205229. PMID 11896603.
- ^ “D-type cyclin-binding regions of proliferating cell nuclear antigen”. J. Biol. Chem. 269 (15): 11030–6. (April 1994). doi:10.1016/S0021-9258(19)78087-9. PMID 7908906.
- ^ a b “Subunit rearrangement of the cyclin-dependent kinases is associated with cellular transformation”. Genes Dev. 7 (8): 1572–83. (August 1993). doi:10.1101/gad.7.8.1572. PMID 8101826.
- ^ “Post-replicative base excision repair in replication foci”. EMBO J. 18 (13): 3834–44. (July 1999). doi:10.1093/emboj/18.13.3834. PMC 1171460. PMID 10393198.
- ^ “A new regulatory motif in cell-cycle control causing specific inhibition of cyclin D/CDK4”. Nature 366 (6456): 704–7. (December 1993). Bibcode: 1993Natur.366..704S. doi:10.1038/366704a0. PMID 8259215.
- ^ a b “Suppression of cell transformation by the cyclin-dependent kinase inhibitor p57KIP2 requires binding to proliferating cell nuclear antigen”. Proc. Natl. Acad. Sci. U.S.A. 95 (4): 1392–7. (February 1998). Bibcode: 1998PNAS...95.1392W. doi:10.1073/pnas.95.4.1392. PMC 19016. PMID 9465025.
- ^ “DNMT1 binds HDAC2 and a new co-repressor, DMAP1, to form a complex at replication foci”. Nat. Genet. 25 (3): 269–77. (July 2000). doi:10.1038/77023. PMID 10888872.
- ^ “PCNA clamp facilitates action of DNA cytosine methyltransferase 1 on hemimethylated DNA”. Genes Cells 7 (10): 997–1007. (October 2002). doi:10.1046/j.1365-2443.2002.00584.x. PMID 12354094.
- ^ “Human DNA-(cytosine-5) methyltransferase-PCNA complex as a target for p21WAF1”. Science 277 (5334): 1996–2000. (September 1997). doi:10.1126/science.277.5334.1996. PMID 9302295.
- ^ “Transcription coactivator p300 binds PCNA and may have a role in DNA repair synthesis”. Nature 410 (6826): 387–91. (March 2001). Bibcode: 2001Natur.410..387H. doi:10.1038/35066610. PMID 11268218.
- ^ “Multivalent interaction of ESCO2 with replication machinery is required for sister chromatid cohesion in vertebrates”. Proc. Natl. Acad. Sci. U.S.A. 117 (2): 1081–1089. (December 2019). doi:10.1073/pnas.1911936117. PMC 6969535. PMID 31879348.
- ^ “The helicase FBH1 is tightly regulated by PCNA via CRL4(Cdt2)-mediated proteolysis in human cells.”. Nucleic Acids Research 41 (13): 6501–13. (Jul 2013). doi:10.1093/nar/gkt397. PMC 3711418. PMID 23677613.
- ^ “Phosphorylation of human Fen1 by cyclin-dependent kinase modulates its role in replication fork regulation”. Oncogene 22 (28): 4301–13. (July 2003). doi:10.1038/sj.onc.1206606. PMID 12853968.
- ^ “Regulation of human flap endonuclease-1 activity by acetylation through the transcriptional coactivator p300”. Mol. Cell 7 (6): 1221–31. (June 2001). doi:10.1016/s1097-2765(01)00272-6. PMID 11430825.
- ^ a b “Regulation of DNA replication and repair proteins through interaction with the front side of proliferating cell nuclear antigen”. EMBO J. 17 (8): 2412–25. (April 1998). doi:10.1093/emboj/17.8.2412. PMC 1170584. PMID 9545252.
- ^ “The DNA repair endonuclease XPG binds to proliferating cell nuclear antigen (PCNA) and shares sequence elements with the PCNA-binding regions of FEN-1 and cyclin-dependent kinase inhibitor p21”. J. Biol. Chem. 272 (39): 24522–9. (September 1997). doi:10.1074/jbc.272.39.24522. PMID 9305916.
- ^ “p21Cip1/Waf1 disrupts the recruitment of human Fen1 by proliferating-cell nuclear antigen into the DNA replication complex”. Proc. Natl. Acad. Sci. U.S.A. 93 (21): 11597–602. (October 1996). Bibcode: 1996PNAS...9311597C. doi:10.1073/pnas.93.21.11597. PMC 38103. PMID 8876181.
- ^ “Interaction of human AP endonuclease 1 with flap endonuclease 1 and proliferating cell nuclear antigen involved in long-patch base excision repair”. Biochemistry 40 (42): 12639–44. (October 2001). doi:10.1021/bi011117i. PMID 11601988.
- ^ a b c “p15(PAF), a novel PCNA associated factor with increased expression in tumor tissues”. Oncogene 20 (4): 484–9. (January 2001). doi:10.1038/sj.onc.1204113. PMID 11313979.
- ^ “Interaction of the p53-regulated protein Gadd45 with proliferating cell nuclear antigen”. Science 266 (5189): 1376–80. (November 1994). Bibcode: 1994Sci...266.1376S. doi:10.1126/science.7973727. PMID 7973727.
- ^ “Direct interaction of Gadd45 with PCNA and evidence for competitive interaction of Gadd45 and p21Waf1/Cip1 with PCNA”. Oncogene 11 (10): 1931–7. (November 1995). PMID 7478510.
- ^ “Characterization of MyD118, Gadd45, and proliferating cell nuclear antigen (PCNA) interacting domains. PCNA impedes MyD118 AND Gadd45-mediated negative growth control”. J. Biol. Chem. 275 (22): 16810–9. (June 2000). doi:10.1074/jbc.275.22.16810. PMID 10828065.
- ^ “Characterisation of the interaction between PCNA and Gadd45”. Oncogene 10 (12): 2427–33. (June 1995). PMID 7784094.
- ^ “Identification of a functional domain in a GADD45-mediated G2/M checkpoint”. J. Biol. Chem. 275 (47): 36892–8. (November 2000). doi:10.1074/jbc.M005319200. PMID 10973963.
- ^ “Interaction of CR6 (GADD45gamma ) with proliferating cell nuclear antigen impedes negative growth control”. J. Biol. Chem. 276 (4): 2766–74. (January 2001). doi:10.1074/jbc.M005626200. PMID 11022036.
- ^ “A novel oncostatin M-inducible gene OIG37 forms a gene family with MyD118 and GADD45 and negatively regulates cell growth”. J. Biol. Chem. 274 (35): 24766–72. (August 1999). doi:10.1074/jbc.274.35.24766. PMID 10455148.
- ^ “Proliferating cell nuclear antigen associates with histone deacetylase activity, integrating DNA replication and chromatin modification”. J. Biol. Chem. 277 (23): 20974–8. (June 2002). doi:10.1074/jbc.M202504200. PMID 11929879.
- ^ “PCNA interacts with hHus1/hRad9 in response to DNA damage and replication inhibition”. Oncogene 19 (46): 5291–7. (November 2000). doi:10.1038/sj.onc.1203901. PMID 11077446.
- ^ “UV-induced binding of ING1 to PCNA regulates the induction of apoptosis”. J. Cell Sci. 114 (Pt 19): 3455–62. (October 2001). doi:10.1242/jcs.114.19.3455. PMID 11682605.
- ^ “A tumor necrosis factor alpha- and interleukin 6-inducible protein that interacts with the small subunit of DNA polymerase delta and proliferating cell nuclear antigen”. Proc. Natl. Acad. Sci. U.S.A. 98 (21): 11979–84. (October 2001). Bibcode: 2001PNAS...9811979H. doi:10.1073/pnas.221452098. PMC 59753. PMID 11593007.
- ^ a b “Chromatin-bound PCNA complex formation triggered by DNA damage occurs independent of the ATM gene product in human cells”. Nucleic Acids Res. 29 (6): 1341–51. (March 2001). doi:10.1093/nar/29.6.1341. PMC 29758. PMID 11239001.
- ^ “Ku antigen, an origin-specific binding protein that associates with replication proteins, is required for mammalian DNA replication”. Biochim. Biophys. Acta 1578 (1–3): 59–72. (October 2002). doi:10.1016/s0167-4781(02)00497-9. PMID 12393188.
- ^ “Regulation of apoptosis and cell cycle progression by MCL1. Differential role of proliferating cell nuclear antigen”. J. Biol. Chem. 275 (50): 39458–65. (December 2000). doi:10.1074/jbc.M006626200. PMID 10978339.
- ^ a b “hMSH3 and hMSH6 interact with PCNA and colocalize with it to replication foci”. Genes Dev. 15 (6): 724–36. (March 2001). doi:10.1101/gad.191201. PMC 312660. PMID 11274057.
- ^ a b “Functional interaction of proliferating cell nuclear antigen with MSH2-MSH6 and MSH2-MSH3 complexes”. J. Biol. Chem. 275 (47): 36498–501. (November 2000). doi:10.1074/jbc.C000513200. PMID 11005803.
- ^ “Human homolog of the MutY repair protein (hMYH) physically interacts with proteins involved in long patch DNA base excision repair”. J. Biol. Chem. 276 (8): 5547–55. (February 2001). doi:10.1074/jbc.M008463200. PMID 11092888.
- ^ a b “A conserved domain of the large subunit of replication factor C binds PCNA and acts like a dominant negative inhibitor of DNA replication in mammalian cells”. EMBO J. 15 (16): 4423–33. (August 1996). doi:10.1002/j.1460-2075.1996.tb00815.x. PMC 452166. PMID 8861969.
- ^ “Towards a proteome-scale map of the human protein-protein interaction network”. Nature 437 (7062): 1173–8. (October 2005). Bibcode: 2005Natur.437.1173R. doi:10.1038/nature04209. PMID 16189514.
- ^ “Human proliferating cell nuclear antigen, poly(ADP-ribose) polymerase-1, and p21waf1/cip1. A dynamic exchange of partners”. J. Biol. Chem. 278 (41): 39265–8. (October 2003). doi:10.1074/jbc.C300098200. PMID 12930846.
- ^ “Structure of the C-terminal region of p21(WAF1/CIP1) complexed with human PCNA”. Cell 87 (2): 297–306. (October 1996). doi:10.1016/s0092-8674(00)81347-1. PMID 8861913.
- ^ “A degradation signal located in the C-terminus of p21WAF1/CIP1 is a binding site for the C8 alpha-subunit of the 20S proteasome”. EMBO J. 20 (10): 2367–75. (May 2001). doi:10.1093/emboj/20.10.2367. PMC 125454. PMID 11350925.
- ^ “Direct interaction of proliferating cell nuclear antigen with the small subunit of DNA polymerase delta”. J. Biol. Chem. 277 (27): 24340–5. (July 2002). doi:10.1074/jbc.M200065200. PMID 11986310.
- ^ “Mediation of proliferating cell nuclear antigen (PCNA)-dependent DNA replication through a conserved p21(Cip1)-like PCNA-binding motif present in the third subunit of human DNA polymerase delta”. J. Biol. Chem. 276 (52): 49258–66. (December 2001). doi:10.1074/jbc.M106990200. PMID 11595739.
- ^ “Identification of a novel protein, PDIP38, that interacts with the p50 subunit of DNA polymerase delta and proliferating cell nuclear antigen”. J. Biol. Chem. 278 (12): 10041–7. (March 2003). doi:10.1074/jbc.M208694200. PMID 12522211.
- ^ “Physical and functional interactions of human DNA polymerase eta with PCNA”. Mol. Cell. Biol. 21 (21): 7199–206. (November 2001). doi:10.1128/MCB.21.21.7199-7206.2001. PMC 99895. PMID 11585903.
- ^ “Stimulation of DNA synthesis activity of human DNA polymerase kappa by PCNA”. Mol. Cell. Biol. 22 (3): 784–91. (February 2002). doi:10.1128/mcb.22.3.784-791.2002. PMC 133560. PMID 11784855.
- ^ “Human DNA polymerase lambda functionally and physically interacts with proliferating cell nuclear antigen in normal and translesion DNA synthesis”. J. Biol. Chem. 277 (50): 48434–40. (December 2002). doi:10.1074/jbc.M206889200. PMID 12368291.
- ^ “Over-expression of human DNA polymerase lambda in E. coli and characterization of the recombinant enzyme”. Genes Cells 7 (7): 639–51. (July 2002). doi:10.1046/j.1365-2443.2002.00547.x. PMID 12081642.
- ^ “A Mammalian bromodomain protein, brd4, interacts with replication factor C and inhibits progression to S phase”. Mol. Cell. Biol. 22 (18): 6509–20. (September 2002). doi:10.1128/mcb.22.18.6509-6520.2002. PMC 135621. PMID 12192049.
- ^ a b “Replication factor C interacts with the C-terminal side of proliferating cell nuclear antigen”. J. Biol. Chem. 272 (3): 1769–76. (January 1997). doi:10.1074/jbc.272.3.1769. PMID 8999859.
- ^ “The DNA-binding subunit p140 of replication factor C is upregulated in cycling cells and associates with G1 phase cell cycle regulatory proteins”. J. Mol. Med. 77 (4): 386–92. (April 1999). doi:10.1007/s001090050365. PMID 10353443.
- ^ a b c “A complex consisting of human replication factor C p40, p37, and p36 subunits is a DNA-dependent ATPase and an intermediate in the assembly of the holoenzyme”. J. Biol. Chem. 272 (30): 18974–81. (July 1997). doi:10.1074/jbc.272.30.18974. PMID 9228079.
- ^ “The subunits of activator 1 (replication factor C) carry out multiple functions essential for proliferating-cell nuclear antigen-dependent DNA synthesis”. Proc. Natl. Acad. Sci. U.S.A. 90 (1): 6–10. (January 1993). Bibcode: 1993PNAS...90....6P. doi:10.1073/pnas.90.1.6. PMC 45588. PMID 8093561.
- ^ “Cloning and characterization of hCTF18, hCTF8, and hDCC1. Human homologs of a Saccharomyces cerevisiae complex involved in sister chromatid cohesion establishment”. J. Biol. Chem. 278 (32): 30051–6. (August 2003). doi:10.1074/jbc.M211591200. PMID 12766176.
- ^ “Polyubiquitination of proliferating cell nuclear antigen by HLTF and SHPRH prevents genomic instability from stalled replication forks”. Proc. Natl. Acad. Sci. U.S.A. 105 (34): 12411–6. (August 2008). Bibcode: 2008PNAS..10512411M. doi:10.1073/pnas.0805685105. PMC 2518831. PMID 18719106.
- ^ “Human HLTF functions as a ubiquitin ligase for proliferating cell nuclear antigen polyubiquitination”. Proc. Natl. Acad. Sci. U.S.A. 105 (10): 3768–73. (March 2008). Bibcode: 2008PNAS..105.3768U. doi:10.1073/pnas.0800563105. PMC 2268824. PMID 18316726.
- ^ “hMMS2 serves a redundant role in human PCNA polyubiquitination”. BMC Mol. Biol. 9: 24. (2008). doi:10.1186/1471-2199-9-24. PMC 2263069. PMID 18284681.
- ^ “Characterisation of the interaction between WRN, the helicase/exonuclease defective in progeroid Werner's syndrome, and an essential replication factor, PCNA”. Mech. Ageing Dev. 124 (2): 167–74. (February 2003). doi:10.1016/s0047-6374(02)00131-8. PMID 12633936.
- ^ “Characterization of the human and mouse WRN 3'-->5' exonuclease”. Nucleic Acids Res. 28 (12): 2396–405. (June 2000). doi:10.1093/nar/28.12.2396. PMC 102739. PMID 10871373.
- ^ “Nucleotide excision repair is associated with the replisome and its efficiency depends on a direct interaction between XPA and PCNA.”. PLOS ONE 7 (11): e49199. (2012). Bibcode: 2012PLoSO...749199G. doi:10.1371/journal.pone.0049199. PMC 3496702. PMID 23152873.
- ^ “XRCC1 co-localizes and physically interacts with PCNA”. Nucleic Acids Res. 32 (7): 2193–201. (2004). doi:10.1093/nar/gkh556. PMC 407833. PMID 15107487.
- ^ “Transcription factor Y-box binding protein 1 binds preferentially to cisplatin-modified DNA and interacts with proliferating cell nuclear antigen”. Cancer Res. 59 (2): 342–6. (January 1999). PMID 9927044.
- ^ “Polyubiquitinated PCNA recruits the ZRANB3 translocase to maintain genomic integrity after replication stress.”. Molecular Cell 47 (3): 396–409. (Aug 10, 2012). doi:10.1016/j.molcel.2012.05.024. PMC 3613862. PMID 22704558.
- ^ “Discrimination of cell cycle phases in PCNA-immunolabeled cells”. BMC Bioinform. 16 (180): 180. (29 May 2015). doi:10.1186/s12859-015-0618-9. PMC 4448323. PMID 26022740.
- ^ “A novel cell permeable DNA replication and repair marker.”. Nucleus (Austin, Tex.) 5 (6): 590–600. (3 September 2014). doi:10.4161/nucl.36290. PMC 4615156. PMID 25484186.
- ^ “PCNA: a silent housekeeper or a potential therapeutic target?”. Trends in Pharmacological Sciences 35 (4): 178–186. (Apr 2014). doi:10.1016/j.tips.2014.02.004. PMID 24655521.
関連文献[編集]
- “Multiple roles of the proliferating cell nuclear antigen: DNA replication, repair and cell cycle control”. 3. (1998). 193–210. doi:10.1007/978-1-4615-5371-7_15. ISBN 978-1-4613-7451-0. PMID 9552415
- “Detection of chromatin-bound PCNA in mammalian cells and its use to study DNA excision repair”. J. Radiat. Res. 40 (1): 1–12. (1999). Bibcode: 1999JRadR..40....1M. doi:10.1269/jrr.40.1. PMID 10408173.
- “Sequence and expression in Escherichia coli of the 40-kDa subunit of activator 1 (replication factor C) of HeLa cells”. Proc. Natl. Acad. Sci. U.S.A. 89 (7): 2516–2520. (1992). Bibcode: 1992PNAS...89.2516C. doi:10.1073/pnas.89.7.2516. PMC 48692. PMID 1313560.
- “Improving responses in hepatomas with circadian-patterned hepatic artery infusions of recombinant interleukin-2”. J. Immunother. 12 (4): 219–223. (1993). doi:10.1097/00002371-199211000-00001. PMID 1477073.
- “Analysis of the proliferating cell nuclear antigen promoter and its response to adenovirus early region 1”. J. Biol. Chem. 265 (27): 16116–25. (1990). doi:10.1016/S0021-9258(17)46196-5. PMID 1975809.
- “Localization of the gene for human proliferating nuclear antigen/cyclin by in situ hybridization”. Hum. Genet. 86 (1): 84–6. (1991). doi:10.1007/bf00205180. PMID 1979311.
- “Structure of the human gene for the proliferating cell nuclear antigen”. J. Biol. Chem. 264 (13): 7466–72. (1989). doi:10.1016/S0021-9258(18)83257-4. PMID 2565339.
- “Human gene for proliferating cell nuclear antigen has pseudogenes and localizes to chromosome 20”. Somat. Cell Mol. Genet. 15 (4): 297–307. (1989). doi:10.1007/BF01534969. PMID 2569765.
- “The cell-cycle regulated proliferating cell nuclear antigen is required for SV40 DNA replication in vitro”. Nature 326 (6112): 471–5. (1987). Bibcode: 1987Natur.326..471P. doi:10.1038/326471a0. PMID 2882422.
- “Cloning and sequence of the human nuclear protein cyclin: homology with DNA-binding proteins”. Proc. Natl. Acad. Sci. U.S.A. 84 (6): 1575–9. (1987). Bibcode: 1987PNAS...84.1575A. doi:10.1073/pnas.84.6.1575. PMC 304478. PMID 2882507.
- “Direct interaction of Gadd45 with PCNA and evidence for competitive interaction of Gadd45 and p21Waf1/Cip1 with PCNA”. Oncogene 11 (10): 1931–7. (1995). PMID 7478510.
- “Lagging strand DNA synthesis at the eukaryotic replication fork involves binding and stimulation of FEN-1 by proliferating cell nuclear antigen”. J. Biol. Chem. 270 (38): 22109–12. (1995). doi:10.1074/jbc.270.38.22109. PMID 7673186.
- “Structure-function relationship of the eukaryotic DNA replication factor, proliferating cell nuclear antigen”. J. Biol. Chem. 270 (38): 22527–34. (1995). doi:10.1074/jbc.270.38.22527. PMID 7673244.
- “A small peptide inhibitor of DNA replication defines the site of interaction between the cyclin-dependent kinase inhibitor p21WAF1 and proliferating cell nuclear antigen”. Curr. Biol. 5 (3): 275–282. (1995). doi:10.1016/S0960-9822(95)00058-3. PMID 7780738.
- “Characterisation of the interaction between PCNA and Gadd45”. Oncogene 10 (12): 2427–33. (1995). PMID 7784094.
- “Construction of a human full-length cDNA bank”. Gene 150 (2): 243–50. (1995). doi:10.1016/0378-1119(94)90433-2. PMID 7821789.
- “D-type cyclin-binding regions of proliferating cell nuclear antigen”. J. Biol. Chem. 269 (15): 11030–6. (1994). doi:10.1016/S0021-9258(19)78087-9. PMID 7908906.
- “Association of proliferating cell nuclear antigen with cyclin-dependent kinases and cyclins in normal and transformed human T lymphocytes”. Blood 84 (10): 3413–21. (1994). doi:10.1182/blood.V84.10.3413.3413. PMID 7949095.
- “Interaction of the p53-regulated protein Gadd45 with proliferating cell nuclear antigen”. Science 266 (5189): 1376–80. (1994). Bibcode: 1994Sci...266.1376S. doi:10.1126/science.7973727. PMID 7973727.
- “The subunits of activator 1 (replication factor C) carry out multiple functions essential for proliferating-cell nuclear antigen-dependent DNA synthesis”. Proc. Natl. Acad. Sci. U.S.A. 90 (1): 6–10. (1993). Bibcode: 1993PNAS...90....6P. doi:10.1073/pnas.90.1.6. PMC 45588. PMID 8093561.
関連項目[編集]
外部リンク[編集]
- PCNA - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス(英語)
- “ANA: Cell cycle related (Mitotic): PCNA type 1 and type 2 Antibody Patterns”. Antibody Patterns.com. 2008年4月15日閲覧。
- Dan Krotz. “Structure of a clamp–loader complex”. Advanced Light Source News. Lawrence Berkeley National Laboratory. 2008年4月15日閲覧。
- Overview of all the structural information available in the PDB for UniProt: P12004 (Proliferating cell nuclear antigen) at the PDBe-KB.